Virtuální karyotyp - Virtual karyotype
Virtuální karyotyp je digitální informace odrážející karyotyp , která je výsledkem analýzy krátkých sekvencí DNA ze specifických lokusů v celém genomu, které jsou izolovány a vyčísleny. Detekuje variace počtu genomických kopií ve vyšším rozlišení pro úroveň než konvenční karyotypizace nebo srovnávací genomová hybridizace na bázi chromozomů (CGH). Hlavní metody používané pro vytváření virtuálních karyotypů jsou genomová hybridizace porovnávající pole a pole SNP .
Pozadí
Karyotypu (obrázek 1) je charakteristická chromozom komplement eukaryotických druhů . Karyotyp je obvykle prezentován jako obraz chromozomů z jedné buňky uspořádané od největší (chromozom 1) po nejmenší (chromozom 22), přičemž pohlavní chromozomy (X a Y) jsou zobrazeny jako poslední. Historicky byly karyotypy získány barvením buněk poté, co byly chemicky zastaveny během buněčného dělení. Karyotypy se používají již několik desetiletí k identifikaci chromozomálních abnormalit v zárodečných i rakovinných buňkách. Konvenční karyotypy mohou posoudit celý genom na změny ve struktuře a počtu chromozomů, ale rozlišení je relativně hrubé, s detekčním limitem 5-10 Mb.

Metoda
Nedávno se objevily platformy pro generování karyotypů s vysokým rozlišením in silico z narušené DNA, jako je pole srovnávací genomové hybridizace (arrayCGH) a pole SNP . Koncepčně jsou pole složena ze stovek až milionů sond, které jsou komplementární k oblasti zájmu v genomu. Rozrušená DNA z testovaného vzorku je fragmentována, označena a hybridizována do pole. Intenzity hybridizačního signálu pro každou sondu jsou používány specializovaným softwarem ke generování log2ratio testu/normálu pro každou sondu v poli. Software, který zná adresu každé sondy v poli a adresu každé sondy v genomu, seřadí sondy v chromozomálním pořadí a rekonstruuje genom in silico (obr. 2 a 3).
Virtuální karyotypy mají dramaticky vyšší rozlišení než konvenční cytogenetika. Skutečné rozlišení bude záviset na hustotě sond na poli. V současné době je Affymetrix SNP6.0 komerčně dostupné pole s nejvyšší hustotou pro aplikace virtuálního karyotypování. Obsahuje 1,8 milionu polymorfních a nepolymorfních markerů pro praktické rozlišení 10–20 kb-přibližně o velikosti genu. To je přibližně 1000krát větší rozlišení než karyotypy získané z konvenční cytogenetiky.
Virtuální karyotypy lze provádět na zárodečných vzorcích pro konstituční poruchy a klinické testování je k dispozici v desítkách laboratoří certifikovaných CLIA ( genetests.org ). Virtuální karyotypizaci lze provést také na čerstvých nebo formalinem fixovaných parafínových nádorech. Mezi laboratoře s certifikací CLIA nabízející testování nádorů patří Creighton Medical Laboratories (vzorky nádorů zalité čerstvým a parafínem) a CombiMatrix Molecular Diagnostics (čerstvé vzorky nádorů).
Různé platformy pro virtuální karyotypování
Karyotypování na bázi pole lze provádět na několika různých platformách, laboratorně vyvinutých i komerčních. Samotná pole mohou být v celém genomu (sondy rozmístěné po celém genomu) nebo cílená (sondy pro genomové oblasti, o nichž je známo, že jsou zapojeny do konkrétního onemocnění) nebo kombinace obou. Dále pole použitá pro karyotypizaci mohou používat nepolymorfní sondy, polymorfní sondy (tj. Obsahující SNP) nebo kombinaci obou. Nepolymorfní sondy mohou poskytovat pouze informace o čísle kopie, zatímco pole SNP mohou poskytovat počet kopií i stav ztráty heterozygotnosti (LOH) v jednom testu. Typy sond používané pro nepolymorfní pole zahrnují cDNA, BAC klony (např. BlueGnome ) a oligonukleotidy (např. Agilent , Santa Clara, CA, USA nebo Nimblegen , Madison, WI, USA). Komerčně dostupná oligonukleotidová pole SNP mohou být na pevné fázi ( Affymetrix , Santa Clara, CA, USA) nebo na bázi kuliček ( Illumina , San Diego, CA, USA). Navzdory rozmanitosti platforem nakonec všichni používají genomovou DNA z rozrušených buněk k opětovnému vytvoření karyotypu s vysokým rozlišením in silico . Konečný produkt zatím nemá jednotný název a byl nazýván virtuální karyotypování, digitální karyotypování, molekulární alelokaryotypování a molekulární karyotypování. Jiné termíny používané k popisu polí použitých pro karyotypizaci zahrnují SOMA (oligonukleotidová mikročipy SNP) a CMA (chromozomová mikročipa). Někteří považují všechny platformy za typ maticové komparativní genomové hybridizace (arrayCGH), zatímco jiní si tento termín vyhrazují pro dvoubarevné metody, a další oddělují pole SNP, protože generují více a odlišných informací než metody dvoubarevného arrayCGH.
Aplikace
Detekce změn počtu kopií
Změny počtu kopií lze vidět na zárodečných i nádorových vzorcích. Změny počtu kopií lze detekovat pomocí polí s nepolymorfními sondami, jako je například arrayCGH, a podle polí založených na SNP. Lidské bytosti jsou diploidní, takže normální počet kopií pro chromozomy bez pohlaví je vždy dva.
- Delece: delece je ztráta genetického materiálu. Delece může být heterozygotní (počet kopií 1) nebo homozygotní (počet kopií 0, nullisomie). Mikrodeletní syndromy jsou příklady konstitučních poruch způsobených malými delecemi v zárodečné DNA. Delece v nádorových buňkách mohou představovat inaktivaci tumor supresorového genu a mohou mít diagnostické, prognostické nebo terapeutické důsledky.
- Zisky: Zisk počtu kopií představuje zisk genetického materiálu. Pokud je zisk pouze z jedné další kopie segmentu DNA, může se tomu říkat duplikace (obr. 4). Pokud existuje ještě jedna kopie celého chromozomu, může se tomu říkat trizomie . Zvýšení počtu kopií ve vzorcích zárodečných linií může být spojeno s onemocněním nebo může být benigní variantou počtu kopií . Když jsou vidět v nádorových buňkách, mohou mít diagnostické, prognostické nebo terapeutické důsledky.
- Zesílení: Technicky je zesílení druh zesílení počtu kopií, ve kterém je počet kopií> 10. V kontextu biologie rakoviny jsou v onkogenech často pozorovány amplifikace . To by mohlo naznačovat horší prognózu, pomoci kategorizovat nádor nebo indikovat způsobilost léčiva. Příkladem způsobilosti léčiva je amplifikace Her2Neu a Herceptin a je poskytnut obraz amplifikace Her2Neu detekované virtuálním karyotypizací pole SNP (obr. 5).
Ztráta heterozygotnosti (LOH), autozygotních segmentů a uniparentální disomie
Autozygotní segmenty a uniparentální disomie (UPD) jsou diploidní/„kopie neutrální“ genetické nálezy, a proto jsou detekovatelné pouze pomocí polí založených na SNP. Jak autozygotní segmenty, tak UPD budou vykazovat ztrátu heterozygotnosti (LOH) s počtem kopií dva karyotypováním pole SNP. Termín Runs of Homozgygosity (ROH) je obecný termín, který lze použít buď pro autozygotní segmenty, nebo pro UPD.
- Autozygotní segment: Autozygotní segment je bi-rodičovský a je vidět pouze v zárodečné linii. Jsou to prodloužené běhy homozygotních markerů v genomu a vyskytují se, když je stejný haplotypový blok zděděn od obou rodičů. Říká se jim také segmenty „ totožné sestupem “ (IBD) a lze je použít k mapování homozygosity.
- Uniparental Disomy: UPD nastává, když jsou obě kopie genu nebo genomové oblasti zděděny od stejného rodiče. To je uniparentální, na rozdíl od autozygotních segmentů, které jsou bi-rodičovské. Pokud jsou přítomny v zárodečné linii, mohou být neškodné nebo mohou souviset s onemocněním, jako jsou Prader-Willi nebo Angelmanův syndrom . Také na rozdíl od autozygotnosti se UPD může vyvinout v nádorových buňkách, což se v literatuře označuje jako získaná UPD nebo kopie neutrální LOH (obr. 6).Získaná UPD je zcela běžná u hematologických i solidních nádorů a uvádí se, že tvoří 20 až 80% LOH pozorovaných u lidských nádorů. Získaná UPD může sloužit jako 2. zásah v hypotéze Knudson Two Hit Hypothesis of Tumorigenesis , a může tedy být biologickým ekvivalentem delece. Protože tento typ léze nelze detekovat pomocí arrayCGH, FISH nebo konvenční cytogenetiky, jsou pro virtuální karyotypizaci nádorů preferována pole založená na SNP.
Obrázek 7 je virtuální karyotyp pole SNP z kolorektálního karcinomu, který ukazuje delece, zisky, zesílení a získanou UPD (kopie neutrální LOH).
Příklady aplikací klinického rakoviny
Virtuální karyotyp lze generovat z téměř jakéhokoli nádoru, ale klinický význam identifikovaných genomových aberací je pro každý typ nádoru odlišný. Klinická užitečnost se liší a vhodnost nejlépe určí onkolog nebo patolog po konzultaci s ředitelem laboratoře laboratoře provádějící virtuální karyotyp. Níže jsou uvedeny příklady typů rakovin, u nichž jsou klinické důsledky specifických genomových aberací dobře prokázány. Tento seznam je reprezentativní, nikoli vyčerpávající. Web pro laboratoř cytogenetiky ve Státní laboratoři hygieny ve Wisconsinu obsahuje další příklady klinicky relevantních genetických změn, které lze snadno zjistit pomocí virtuálního karyotypování. [1]
Neuroblastom
Na základě série 493 vzorků neuroblastomu bylo hlášeno, že celkový genomový vzorec, testovaný karyotypováním založeným na poli, je prediktorem výsledku u neuroblastomu:
- Nádory vykazující výhradně změny počtu kopií celého chromozomu byly spojeny s vynikajícím přežitím.
- Nádory vykazující jakýkoli druh změn počtu kopií segmentálních chromozomů byly spojeny s vysokým rizikem relapsu.
- U nádorů vykazujících segmentální změny byly dalšími nezávislými prediktory sníženého celkového přežití amplifikace MYCN, delece 1p a 11q a zisk 1q.
Dřívější publikace rozdělovaly neuroblastomy na tři hlavní podtypy na základě cytogenetických profilů:
- Podtyp 1: příznivý neuroblastom s téměř triploidií a převahou numerických zisků a ztrát, většinou představující nemetastatické stadia 1, 2 a 4S NB.
- Podtypy 2A a 2B: nalezeny u nepříznivého rozšířeného neuroblastomu, stádia 3 a 4, se ztrátou 11q a ziskem 17q bez amplifikace MYCN (podtyp 2A) nebo se zesílením MYCN často společně s delecemi 1p a ziskem 17q (podtyp 2B).
Wilmsův nádor
Tumorově specifická ztráta heterozygotnosti (LOH) pro chromozomy 1p a 16q identifikuje podskupinu pacientů s Wilmsovým nádorem, kteří mají významně zvýšené riziko relapsu a smrti. LOH pro tyto chromozomální oblasti lze nyní použít jako nezávislý prognostický faktor společně se stadiem onemocnění k zaměření intenzity léčby na riziko selhání léčby.
Karcinom renálních buněk
Novotvary epitelu ledvin mají charakteristické cytogenetické aberace, které mohou pomoci při klasifikaci. Viz také Atlas genetiky a cytogenetiky v onkologii a hematologii .
- Čirý buněčný karcinom: ztráta 3 p
- Papilární karcinom: trizomie 7 a 17
- Chromofobní karcinom: hypodiploid se ztrátou chromozomů 1, 2, 6, 10, 13, 17, 21
Karyotypování na bázi pole lze použít k identifikaci charakteristických chromozomálních aberací u renálních nádorů s náročnou morfologií. Karyotypování na poli funguje dobře u nádorů zapuštěných do parafinu a je přístupné rutinnímu klinickému použití.
Nedávná literatura navíc uvádí, že určité chromozomální aberace jsou spojeny s výsledkem ve specifických podtypech renálních epiteliálních nádorů.
Renální karcinom z jasných buněk: del 9p a del 14q jsou špatné prognostické ukazatele.
Papilární karcinom ledvinových buněk: duplikace 1q značí fatální progresi.
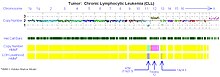
Chronická lymfocytární leukémie
Karyotypování založené na poli je nákladově efektivní alternativou k FISH pro detekci chromozomálních abnormalit u chronické lymfocytární leukémie (CLL). Několik studií klinické validace prokázalo> 95% shodu se standardním panelem CLL FISH. Kromě toho mnoho studií využívajících karyotypování založených na poli identifikovalo „atypické delece“, které standardní sondy FISH vynechaly, a získaly uniparentální disomii v klíčových lokusech pro prognostické riziko u CLL.
V buňkách CLL jsou rozpoznávány čtyři hlavní genetické aberace, které mají zásadní vliv na chování chorob.
- Zvláště škodlivé jsou delece části krátkého ramene chromozomu 17 (del 17p), které cílí na p53. Pacienti s touto abnormalitou mají výrazně kratší interval, než vyžadují terapii, a kratší přežití. Tato abnormalita se vyskytuje u 5–10% pacientů s CLL.
- Delece dlouhého ramene na chromozomu 11 (del 11q) jsou také nepříznivé, i když ne v takové míře, jako je tomu u del 17p. Abnormalita se zaměřuje na gen ATM a vyskytuje se zřídka u CLL (5–10%).
- Trisomie 12, další chromozom 12, je poměrně častým nálezem vyskytujícím se u 20–25% pacientů a poskytuje střední prognózu.
- Delece 13q14 (del 13q14) je nejčastější abnormalitou u CLL se zhruba 50% pacientů s buňkami obsahujícími tento defekt. Když je del 13q14 viděn izolovaně, pacienti mají nejlepší prognózu a většina bude žít mnoho let, dokonce desetiletí, bez nutnosti terapie.
Mnohočetný myelom
Avet-Loiseau a kol. v Journal of Clinical Oncology použil karyotypizaci pole SNP 192 vzorků mnohočetného myelomu (MM) k identifikaci genetických lézí spojených s prognózou, které byly poté validovány v samostatné kohortě (n = 273). U MM činí nedostatek proliferativního klonu konvenční cytogenetiku informativní pouze v ~ 30% případů. Panely FISH jsou užitečné v MM, ale standardní panely by nezjistily několik klíčových genetických abnormalit uvedených v této studii.
- Virtuální karyotypizace identifikovala chromozomální abnormality v 98% případů MM
- del (12p13.31) je nezávislý nepříznivý marker
- amp (5q31.1) je výhodný marker
- Prognostický dopad amp (5q31.1) převyšuje hyperdiploidii a také identifikuje pacienty, kteří mají z léčby vysokými dávkami velký prospěch.
Karyotypování založené na poli nemůže detekovat vyvážené translokace, jako je t (4; 14) pozorované u ~ 15% MM. FISH pro tuto translokaci by proto mělo být provedeno také v případě, že k detekci změn počtu kopií celého genomu prognostického významu v MM pomocí polí SNP.
Meduloblastom
Karyotypizace 260 meduloblastomů na bázi pole od Pfister S, et al. vedly k následujícím klinickým podskupinám na základě cytogenetických profilů:
- Špatná prognóza: zisk 6q nebo zesílení MYC nebo MYCN
- Střední: zisk 17q nebo i (17q) bez zisku 6q nebo zesílení MYC nebo MYCN
- Vynikající prognóza: 6q a 17q vyvážené nebo 6q delece
Oligodendrogliom
Společná delece 1p/19q je považována za "genetický podpis" oligodendrogliomu . Alelické ztráty na 1p a 19q, buď samostatně nebo v kombinaci, jsou běžnější u klasických oligodendrogliomů než u astrocytomů nebo oligoastrocytomů. V jedné studii klasický oligodendrogliom vykazoval ztrátu 1 p v 35 ze 42 (83%) případů, 19q ztrátu ve 28 z 39 (72%) a tyto byly kombinovány v 27 z 39 (69%) případů; mezi low-grade a anaplastickými oligodendrogliomy nebyl žádný významný rozdíl ve ztrátě heterozygotnosti 1p/19q. Delece 1p/19q byla korelována jak s chemosenzitivitou, tak se zlepšenou prognózou u oligodendrogliomů. Většina větších center pro léčbu rakoviny rutinně kontroluje deleci 1p/19q jako součást patologické zprávy pro oligodendrogliomy. Stav lokusů 1p/19q lze detekovat pomocí FISH nebo virtuálního karyotypování. Virtuální karyotypizace má tu výhodu, že se vyhodnotí celý genom v jednom testu, stejně jako lokusy 1p/19q. To umožňuje posouzení dalších klíčových lokusů u gliových nádorů, jako je stav počtu kopií EGFR a TP53.
Zatímco prognostický význam delecí 1p a 19q je dobře zaveden u anaplastických oligodendrogliomů a smíšených oligoastrocytomů, prognostický význam delecí pro gliomy nízkého stupně je kontroverznější. Pokud jde o gliomy nízkého stupně, nedávná studie také naznačuje, že ko-delece 1p/19q může být spojena s translokací (1; 19) (q10; p10), která, stejně jako kombinovaná delece 1p/19q, je spojena s vyšší celkové přežití a přežití bez progrese u pacientů s nízkým stupněm gliomu. Oligodendrogliomy vykazují pouze zřídka mutace v genu p53, což je na rozdíl od jiných gliomů. Amplifikace receptoru epidermálního růstového faktoru a celá kodexie 1p/19q se vzájemně vylučují a predikují zcela odlišné výsledky, přičemž amplifikace EGFR předpovídá špatnou prognózu.
Glioblastom
Yin a kol. studovali 55 glioblastomových a 6 GBM buněčných linií pomocí karyotypování pole SNP. Získaná UPD byla identifikována na 17 p ve 13/61 případech. Významně zkrácená doba přežití byla nalezena u pacientů s delecí 13q14 (RB) nebo delecí/získanou UPD 17p13.1 (p53). Dohromady tyto výsledky naznačují, že tato technika je rychlou, robustní a levnou metodou k profilování abnormalit celého genomu v GBM. Protože karyotypizaci pole SNP lze provádět na nádorech zalitých v parafinu, je to atraktivní volba v případě, že nádorové buňky nerostou v kultuře pro metafázovou cytogenetiku, nebo když se po fixaci vzorku formalinem vyvine touha po karyotypizaci.
Důležitost detekce získané UPD (kopie neutrální LOH) u glioblastomu:
- U pacientů s abnormalitou 17p bylo ~ 50% delecí a ~ 50% aUPD
- 17p del i 17p UPD byly spojeny s horším výsledkem.
- 9/13 mělo homozygotní mutace TP53, které byly základem 17p UPD.
Navíc v případech s nejistým stupněm podle morfologie může při diagnostice pomoci genomové profilování.
- Souběžný zisk 7 a ztráta 10 je v podstatě patognomický pro GBM
- K amplifikaci EGFR, ztrátě PTEN (na 10q) a ztrátě p16 (na 9p) dochází téměř výhradně u glioblastomu a může poskytnout prostředky k odlišení anaplastického astrocytomu od glioblastomu.
Akutní lymfoblastická leukémie
Cytogenetika , studium charakteristických velkých změn v chromozomech z nádorových buněk , je stále větší pozornost jako významný prediktor výsledku v akutní lymfoblastické leukémie (ALL).
Poznámka: Vyvážené translokace nelze detekovat karyotypováním založeným na poli (viz Omezení níže).
Některé cytogenetické podtypy mají horší prognózu než jiné. Tyto zahrnují:
- Translokace mezi chromozomy 9 a 22, známá jako chromozom Philadelphia , se vyskytuje asi u 20% dospělých a 5% u pediatrických případů ALL.
- K translokaci mezi chromozomy 4 a 11 dochází asi ve 4% případů a je nejčastější u kojenců mladších 12 měsíců.
- Ne všechny translokace chromozomů mají horší prognózu. Některé translokace jsou relativně příznivé. Hyperdiploidie (> 50 chromozomů) je například dobrým prognostickým faktorem.
- Hodnocení změn počtu kopií v celém genomu lze provést konvenční cytogenetikou nebo virtuálním karyotypováním. Virtuální karyotypování pole SNP může detekovat změny počtu kopií a stav LOH, zatímco arrayCGH dokáže detekovat pouze změny počtu kopií. Kopie neutrální LOH (získaná uniparentální disomie) byla hlášena na klíčových lokusech u ALL, jako je gen CDKN2A při 9 p, které mají prognostický význam. Virtuální karyotypování pole SNP může snadno detekovat kopie neutrální LOH. Pole CGH, FISH a konvenční cytogenetika nemohou detekovat kopie neutrální LOH.
| Cytogenetická změna | Kategorie rizika |
|---|---|
| Chromozom Philadelphia | Špatná prognóza |
| t (4; 11) (q21; q23) | Špatná prognóza |
| t (8; 14) (q24,1; q32) | Špatná prognóza |
| Komplexní karyotyp (více než čtyři abnormality) | Špatná prognóza |
| Nízká hypodiploidie nebo téměř triploidie | Špatná prognóza |
| Vysoká hyperdiploidie | Dobrá prognóza |
| del (9p) | Dobrá prognóza |
Korelace prognózy s cytogenetickým nálezem kostní dřeně u akutní lymfoblastické leukémie
| Prognóza | Cytogenetické nálezy |
|---|---|
| Příznivý | Hyperdiploidie> 50; t (12; 21) |
| středně pokročilí | Hyperdioloidie 47 -50; Normální (diploidie); del (6q); Přeuspořádání 8q24 |
| Nepříznivý | Hypodiploidy-téměř haploidy; Blízko tetraploidie; del (17p); t (9; 22); t (11q23) |
Neklasifikovaná ALL je považována za zprostředkovanou prognózu.
Myelodysplastický syndrom
Myelodysplastický syndrom (MDS) má pozoruhodnou klinickou, morfologickou a genetickou heterogenitu. Cytogenetika hraje rozhodující roli v Mezinárodním systému prognostického skóre (IPSS) Světové zdravotnické organizace pro MDS.
- Dobrá prognóza: normální karyotyp, izolovaný del (5q), izolovaný del (20q), -Y
- Špatná prognóza: komplexní abnormality (tj.> = 3 abnormality), −7 nebo del (7q)
- Mezilehlá prognóza: všechny ostatní abnormality, včetně trizomie 8 a del (11q)
Při srovnání cytogenetiky metafáze, panelu FISH a karyotypování pole SNP pro MDS bylo zjištěno, že každá technika poskytuje podobný diagnostický výtěžek. Žádná jednotlivá metoda nezjistila všechny defekty a míra detekce se zlepšila o ~ 5%, když byly použity všechny tři metody.
Získaná UPD, která není detekovatelná FISH nebo cytogenetikou, byla hlášena na několika klíčových lokusech v MDS pomocí karyotypování pole SNP, včetně delece 7/7q.
Myeloproliferativní novotvary/myeloproliferativní poruchy
Philadelphia chromozom-negativní myeloproliferativní novotvary (MPN) včetně polycythemia vera, esenciální trombocytémie a primární myelofibrózy vykazují inherentní tendenci k transformaci na leukémii (MPN-blast fáze), která je doprovázena získáváním dalších genomových lézí. Ve studii se 159 případy byla analýza pole SNP schopna zachytit prakticky všechny cytogenetické abnormality a odhalit další léze s potenciálně důležitými klinickými důsledky.
- Počet genomových alterací byl ve blastické fázi více než 2 až 3krát vyšší než v chronické fázi onemocnění.
- Delece 17p (TP53) byla významně spojena s předchozí expozicí hydroxymočovině a také komplexním karyotypem ve vzorcích s MPN-blastickou krizí. Delece i 17p kopie neutrální LOH byly spojeny se složitým karyotypem, špatným prognostickým markerem u myeloidních malignit. Kopie neutrální LOH (získaná UPD) je snadno detekovatelná karyotypem pole SNP, ale nikoli cytogenetikou, FISH nebo maticí CGH.
- Pacienti v blastické fázi se ztrátou chromozomálního materiálu na 7q vykazovali špatné přežití. Je známo, že ztráta 7q je prediktivní pro rychlou progresi a špatnou odpověď v terapii AML. Pacienti s MPN-blastovou fází s cytogeneticky nezjistitelným 7q kopií neutrálního LOH měli srovnatelnou míru přežití s pacienty se 7/7q ve svých leukemických buňkách.
- 9p kopie neutrální LOH s homozygotní mutací JAK2 byla také spojena s horším výsledkem v MPN-blast krizi ve srovnání s pacienty buď s heterozygotní JAK2V617F nebo divokým typem JAK2. Na rozdíl od LOH na 17p byl prognostický dopad 9pCNN-LOH nezávislý na zavedených rizikových faktorech, jako je 7/7q, 5q nebo komplexní karyotyp.
Kolorektální karcinom
Identifikace biomarkerů u kolorektálního karcinomu je zvláště důležitá u pacientů s onemocněním stádia II, kde u méně než 20% dochází k recidivě tumoru. 18q LOH je zavedený biomarker spojený s vysokým rizikem recidivy tumoru ve stadiu II rakoviny tlustého střeva. Obrázek 7 ukazuje karyotyp pole SNP kolorektálního karcinomu (pohled celého genomu).
Kolorektální rakoviny jsou klasifikovány do specifických fenotypů nádorů na základě molekulárních profilů, které mohou být integrovány s výsledky dalších pomocných testů, jako je testování nestability mikrosatelitů, IHC a stav mutace KRAS:
- Chromozomální nestabilita (CIN), která má alelickou nerovnováhu v řadě chromozomálních lokusů, včetně 5q, 8p, 17p a 18q (obr. 7).
- Mikrosatelitní nestabilita (MSI), která mívá diploidní karyotypy.
Zhoubné rhabdoidní nádory
Zhoubné rhabdoidní nádory jsou vzácné, vysoce agresivní novotvary, které se nejčastěji vyskytují u kojenců a malých dětí. Vzhledem k jejich heterogenním histologickým rysům může být diagnostika často obtížná a může dojít k nesprávné klasifikaci. V těchto nádorech funguje gen INI1 (SMARCB1) na chromozomu 22q jako klasický tumor supresorový gen. K deaktivaci INI1 může dojít prostřednictvím delece, mutace nebo získané UPD.
V nedávné studii karyotypizace pole SNP identifikovala delece nebo LOH 22q u 49/51 rhabdoidních nádorů. Z nich bylo 14 kopií neutrálních LOH (nebo získaných UPD), které jsou detekovatelné karyotypizací pole SNP, ale nikoli pomocí FISH, cytogenetiky nebo arrayCGH. MLPA detekovalo jeden exon homozygotní deleci v jednom vzorku, který byl pod rozlišením pole SNP.
Karyotypizaci SNP pole lze použít například k rozlišení meduloblastomu s izochromozomem 17q od primárního rhabdoidního tumoru se ztrátou 22q11.2. Pokud je to uvedeno, pak může být použita molekulární analýza INI1 pomocí MLPA a přímého sekvenování. Jakmile jsou nalezeny změny související s nádorem, lze provést analýzu zárodečné DNA od pacienta a rodičů, aby se vyloučila dědičná nebo de novo zárodečná mutace nebo delece INI1, aby bylo možné provést odpovídající hodnocení rizika recidivy.
Uveální melanom
Nejdůležitější genetickou změnou spojenou se špatnou prognózou u uveálního melanomu je ztráta celé kopie chromozomu 3 ( monosomie 3), která silně koreluje s metastatickým šířením. Zisky na chromozomech 6 a 8 se často používají k upřesnění prediktivní hodnoty obrazovky Monosomy 3, přičemž zisk 6p indikuje lepší prognózu a zisk 8q indikuje horší prognózu u nádorů disomy 3. Ve vzácných případech mohou nádory monosomie 3 duplikovat zbývající kopii chromozomu, aby se vrátily do disomického stavu označovaného jako izodisomie . Izodisomie 3 je prognosticky ekvivalentní monosomii 3 a obě mohou být detekovány testy na ztrátu heterozygotnosti chromozomu 3 .
Omezení
Na rozdíl od karyotypů získaných z konvenční cytogenetiky jsou virtuální karyotypy rekonstruovány počítačovými programy pomocí signálů získaných z narušené DNA. V podstatě počítačový program opraví translokace, když seřadí signály v chromozomálním pořadí. Virtuální karyotypy proto nemohou detekovat vyvážené translokace a inverze . Mohou také detekovat pouze genetické aberace v oblastech genomu, které jsou reprezentovány sondami v poli. Virtuální karyotypy navíc generují relativní počet kopií normalizovaný proti diploidnímu genomu, takže tetraploidní genomy budou kondenzovány do diploidního prostoru, pokud nebude provedena renormalizace. Renormalizace vyžaduje doplňkový buněčný test, jako je FISH, pokud jeden používá arrayCGH. U karyotypů získaných z polí na bázi SNP lze tetraploidii často odvodit ze zachování heterozygotnosti v oblasti zjevné ztráty počtu kopií. Nízkoúrovňový mozaismus nebo malé subklony nemusí být detekovány virtuálními karyotypy, protože přítomnost normálních buněk ve vzorku tlumí signál z abnormálního klonu. Přesný bod selhání, pokud jde o minimální procento neoplastických buněk, bude záviset na konkrétní platformě a použitých algoritmech. Mnoho softwarových programů pro analýzu počtu kopií použitých ke generování karyotypů založených na poli bude váhat s méně než 25–30% nádorových/abnormálních buněk ve vzorku. V onkologických aplikacích však lze toto omezení minimalizovat strategiemi obohacování nádorů a softwarem optimalizovaným pro použití s onkologickými vzorky. Analytické algoritmy se rychle vyvíjejí a některé jsou dokonce navrženy tak, aby prospívaly „normální klonovou kontaminací“, takže se předpokládá, že toto omezení se bude nadále rozptylovat.
Viz také
- DECIPHER , databáze chromozomální nerovnováhy a fenotypu u lidí využívající zdroje Ensembl