Sekvenční zarovnání - Sequence alignment
V bioinformatice je seřazení sekvencí způsob uspořádání sekvencí DNA , RNA nebo proteinu k identifikaci oblastí podobnosti, které mohou být důsledkem funkčních, strukturálních nebo evolučních vztahů mezi sekvencemi. Zarovnané sekvence nukleotidových nebo aminokyselinových zbytků jsou typicky znázorněny jako řádky v matici . Mezery jsou vloženy mezi zbytky tak, aby stejné nebo podobné znaky byly zarovnány do po sobě jdoucích sloupců. Sekvenční zarovnání se také používají pro nebiologické sekvence, jako je výpočet nákladů na vzdálenost mezi řetězci v přirozeném jazyce nebo ve finančních datech.

Sekvence jsou aminokyseliny pro zbytky 120-180 proteinů. Zbytky, které jsou konzervovány napříč všemi sekvencemi, jsou zvýrazněny šedě. Pod sekvencemi proteinů je klíč označující konzervovanou sekvenci (*), konzervativní mutace (:), semi-konzervativní mutace (.) A nekonzervativní mutace ().
Výklad
Pokud dvě sekvence v zarovnání sdílejí společného předka, neshody mohou být interpretovány jako bodové mutace a mezery jako indely (tj . Inzerční nebo deleční mutace) zavedené do jedné nebo obou linií v čase, protože se od sebe navzájem lišily. Při sekvenčním seřazení proteinů lze stupeň podobnosti mezi aminokyselinami zaujímajícími určitou pozici v sekvenci interpretovat jako hrubé měřítko toho, jak konzervativní je konkrétní oblast nebo motiv sekvence mezi liniemi. Absence substitucí nebo přítomnost pouze velmi konzervativních substitucí (to znamená substituce aminokyselin, jejichž postranní řetězce mají podobné biochemické vlastnosti) v konkrétní oblasti sekvence, naznačují, že tato oblast má strukturální nebo funkční význam. Ačkoli nukleotidové báze DNA a RNA jsou si navzájem více podobné než aminokyseliny, zachování párů bází může naznačovat podobnou funkční nebo strukturní roli.
Vyrovnávací metody
Velmi krátké nebo velmi podobné sekvence lze zarovnat ručně. Většina nejzajímavějších problémů však vyžaduje zarovnání dlouhých, vysoce variabilních nebo extrémně početných sekvencí, které nelze zarovnat pouze lidským úsilím. Místo toho jsou lidské znalosti aplikovány při konstrukci algoritmů k produkci vysoce kvalitních zarovnání sekvencí a příležitostně při úpravě konečných výsledků tak, aby odrážely vzorce, které je obtížné algoritmicky znázornit (zejména v případě nukleotidových sekvencí). Výpočetní přístupy k zarovnání sekvence obecně spadají do dvou kategorií: globální zarovnání a místní zarovnání . Výpočet globálního zarovnání je forma globální optimalizace, která „nutí“ zarovnání pokrýt celou délku všech sekvencí dotazů. Naproti tomu lokální zarovnání identifikuje oblasti podobnosti v rámci dlouhých sekvencí, které jsou celkově často velmi odlišné. Místní zarovnání jsou často vhodnější, ale může být obtížnější je vypočítat kvůli další výzvě při identifikaci oblastí podobnosti. Na problém zarovnání sekvence byla použita řada výpočetních algoritmů. Patří sem pomalé, ale formálně správné metody, jako je dynamické programování . Patří sem také účinné heuristické algoritmy nebo pravděpodobnostní metody určené pro rozsáhlé vyhledávání v databázi, které nezaručují nalezení nejlepších shod.
Zastoupení
Zarovnání jsou běžně reprezentována jak graficky, tak v textovém formátu. Téměř ve všech reprezentacích zarovnání sekvence jsou sekvence zapsány do řádků uspořádaných tak, že zarovnané zbytky se objevují v po sobě jdoucích sloupcích. V textových formátech jsou zarovnané sloupce obsahující identické nebo podobné znaky označeny systémem symbolů zachování. Stejně jako na obrázku výše je k zobrazení identity mezi dvěma sloupci použit symbol hvězdičky nebo potrubí; další méně obvyklé symboly zahrnují dvojtečku pro konzervativní substituce a tečku pro semikonzervativní substituce. Mnoho programů vizualizace sekvencí také používá barvu k zobrazení informací o vlastnostech jednotlivých prvků sekvence; v sekvencích DNA a RNA to odpovídá přiřazení každého nukleotidu vlastní barvy. Při zarovnání proteinů, jako je to na obrázku výše, se často používá barva k označení vlastností aminokyselin, což pomáhá při posuzování zachování dané substituce aminokyseliny. U více sekvencí je poslední řádek v každém sloupci často konsensuální sekvencí určenou seřazením; konsensuální sekvence je také často znázorněna v grafickém formátu s logem sekvence, ve kterém velikost každého nukleotidu nebo písmena aminokyseliny odpovídá jeho stupni zachování.
Sekvenční zarovnání lze uložit do široké škály formátů souborů založených na textu, z nichž mnohé byly původně vyvinuty ve spojení s konkrétním programem zarovnání nebo implementací. Většina webových nástrojů umožňuje omezený počet vstupních a výstupních formátů, například formát FASTA a formát GenBank, a výstup nelze snadno upravovat. Několik konverzní programy, které poskytují grafické a / nebo rozhraní příkazové řádky jsou k dispozici, jako je READSEQ a EMBOSS . Existuje také několik programovacích balíčků, které tuto funkci převodu poskytují, například BioPython , BioRuby a BioPerl . Soubory SAM/BAM používají řetězecový formát CIGAR (Compact Idiosyncratic Gapped Alignment Report) k reprezentaci zarovnání sekvence k odkazu kódováním sekvence událostí (např. Shoda/nesoulad, vložení, odstranění).
Formát CIGAR
Ref. : GTCGTAGAATA
Čtení : CACGTAG — TA
CIGAR: 2S5M2D2M kde:
2S = 2 měkké výstřižky (může to být nesoulad nebo čtení delší než odpovídající sekvence)
5M = 5 odpovídá nebo nesouhlasí
2D = 2 vymazání
2M = 2 shody nebo neshody
Původní formát CIGAR z programu zarovnání ospravedlnění nerozlišoval mezi neshodami nebo shodami se znakem M.
Specifikační dokument SAMv1 definuje novější kódy CIGAR. Ve většině případů je výhodnější použít znaky '=' a 'X' k označení shod nebo nesouladů než starší znak 'M', který je nejednoznačný.
| Kód doutníku | BAM celé číslo | Popis | Konzumuje dotaz | Spotřebovává reference |
| M | 0 | zarovnání shody (může jít o sekvenční shodu nebo nesoulad) | Ano | Ano |
| Já | 1 | vložení do odkazu | Ano | Ne |
| D | 2 | vymazání z odkazu | Ne | Ano |
| N. | 3 | přeskočená oblast z reference | Ne | Ano |
| S | 4 | soft clipping (ořezané sekvence přítomné v SEQ) | Ano | Ne |
| H | 5 | tvrdé výstřižky (ořezané sekvence NEJSOU přítomny v SEQ) | Ne | Ne |
| P | 6 | padding (tiché vymazání z polstrované reference) | Ne | Ne |
| = | 7 | sekvenční shoda | Ano | Ano |
| X | 8 | neshoda sekvencí | Ano | Ano |
- „Spotřebovává dotaz“ a „spotřebovává referenci“ udává, zda operace CIGAR způsobí, že zarovnání postupuje po sekvenci dotazu respektive referenční sekvenci.
- H může být přítomen pouze jako první a/nebo poslední operace.
- S může mít pouze operace H mezi nimi a konci řetězce CIGAR.
- Pro zarovnání mRNA k genomu představuje operace N intron. U ostatních typů zarovnání není interpretace N definována.
- Součet délek operací M/I/S/=/X se musí rovnat délce SEQ
Globální a místní sladění
Globální zarovnání, která se pokoušejí zarovnat každý zbytek v každé sekvenci, jsou nejužitečnější, když jsou sekvence v sadě dotazů podobné a mají zhruba stejnou velikost. (To neznamená, že globální zarovnání nemůže začínat a/nebo končit mezerami.) Obecnou technikou globálního zarovnání je algoritmus Needleman – Wunsch , který je založen na dynamickém programování. Místní zarovnání jsou užitečnější pro nepodobné sekvence, u nichž je podezření, že obsahují oblasti podobnosti nebo podobné motivy sekvence v kontextu jejich větší sekvence. Algoritmus Smith-Waterman je obecná místní způsob vyrovnání založeno na stejném dynamické programování systému, ale s dalšími možnostmi začínat a končit v libovolném místě.
Hybridní metody, známé jako semi-globální nebo „glokální“ (zkratka pro Glo bal-lo cal ) metody, hledat nejlepší možné částečné seřazení dvou sekvencí (jinými slovy, kombinace jednoho nebo obou startů a jeden nebo oba konce jsou uvedeny jako zarovnané). To může být zvláště užitečné, když se downstream část jedné sekvence překrývá s upstream částí druhé sekvence. V tomto případě není ani globální, ani místní zarovnání zcela vhodné: globální zarovnání by se pokusilo vynutit zarovnání tak, aby přesahovalo oblast překrytí, zatímco místní zarovnání nemusí zcela překrývat oblast překrytí. Další případ, kdy je užitečné pologlobální zarovnání, je, když je jedna sekvence krátká (například sekvence genu) a druhá je velmi dlouhá (například sekvence chromozomů). V takovém případě by měla být krátká sekvence zarovnána globálně (plně), ale pro dlouhou sekvenci je požadováno pouze lokální (částečné) zarovnání.
Rychlá expanze genetických dat zpochybňuje rychlost současných algoritmů zarovnání sekvencí DNA. Základní potřeby efektivní a přesné metody pro objevování variant DNA vyžadují inovativní přístupy pro paralelní zpracování v reálném čase. Optické výpočetní přístupy byly navrženy jako slibné alternativy současných elektrických implementací, jejich použitelnost je však ještě třeba testovat [1] .
Párové zarovnání
K vyhledání nejlépe odpovídajících kusových (lokálních nebo globálních) zarovnání dvou sekvencí dotazu se používají metody párování sekvencí. Párová zarovnání lze použít pouze mezi dvěma sekvencemi najednou, ale jsou efektivní pro výpočet a často se používají pro metody, které nevyžadují extrémní přesnost (například prohledávání sekvence v databázi s vysokou podobností s dotazem). Tři primární metody vytváření párových zarovnání jsou metody s bodovou maticí, dynamické programování a slovní metody; avšak techniky zarovnání více sekvencí mohou také zarovnat páry sekvencí. Ačkoli každá metoda má své individuální silné a slabé stránky, všechny tři párové metody mají potíže s vysoce opakujícími se sekvencemi s nízkým obsahem informací - zejména tam, kde se počet opakování liší ve dvou sekvencích, které mají být sladěny.
Maximální jedinečná shoda
Jedním ze způsobů, jak kvantifikovat užitečnost daného párového zarovnání, je „ maximální jedinečná shoda “ (MUM) nebo nejdelší subsekvence, která se vyskytuje v obou sekvencích dotazů. Delší sekvence MUM obvykle odrážejí bližší příbuznost. ve vícenásobné porovnání sekvencí z genomů ve výpočetní biologii . Identifikace MUM a dalších potenciálních kotev je prvním krokem ve větších zarovnávacích systémech, jako je MUMmer . Kotvy jsou oblasti mezi dvěma genomy, kde jsou velmi podobné. Abychom pochopili, co je MUM, můžeme rozdělit každé slovo ve zkratce. Shoda znamená, že se podřetězec vyskytuje v obou sekvencích, které mají být zarovnány. Unikátní znamená, že se podřetězec v každé sekvenci vyskytuje pouze jednou. Nakonec maximální uvádí, že podřetězec není součástí jiného většího řetězce, který splňuje oba předchozí požadavky. Hlavní myšlenkou je, že dlouhé sekvence, které se přesně shodují a vyskytují se pouze jednou v každém genomu, jsou téměř jistě součástí globálního uspořádání.
Přesněji:
"Vzhledem ke dvěma genomům A a B je podřetězec Maximal Unique Match (MUM) běžným podřetězem A a B o délce delší než stanovená minimální délka d (standardně d = 20) tak, že
- je maximální, to znamená, že jej nelze prodloužit na obou koncích, aniž by došlo k nesouladu; a
- je jedinečný v obou sekvencích “
Metody bodové matice

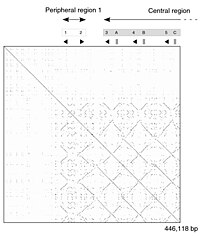
DNA dot pozemek o lidské zinkový prst transkripčního faktoru (GenBank číslo NM_002383), ukazující regionální self-podoba . Hlavní úhlopříčka představuje zarovnání sekvence sama se sebou; čáry mimo hlavní úhlopříčku představují podobné nebo opakující se vzory v sekvenci. Toto je typický příklad opakování zápletky .
|
Dot-matrix přístup, který implicitně vytváří rodinu zarovnání pro jednotlivé sekvenční oblasti, je kvalitativní a koncepčně jednoduchý, i když časově náročný na analýzu ve velkém měřítku. Při absenci šumu lze snadno určit určité sekvenční funkce-například vložení, vymazání, opakování nebo obrácené opakování- z bodové matice. Pro konstrukci grafu s bodovou maticí jsou tyto dvě sekvence zapsány podél horního řádku a levého sloupce dvojrozměrné matice a tečka je umístěna v libovolném bodě, kde se shodují znaky v příslušných sloupcích-toto je typický graf opakování . Některé implementace mění velikost nebo intenzitu tečky v závislosti na stupni podobnosti těchto dvou znaků, aby vyhovovaly konzervativním substitucím. Tečkové grafy velmi úzce souvisejících sekvencí se zobrazí jako jedna čára podél hlavní úhlopříčky matice .
Mezi problémy s bodovými grafy jako technikou zobrazení informací patří: šum, nejasnost, neintuitivnost, potíže s extrahováním souhrnných statistik zápasů a pozic zápasů na těchto dvou sekvencích. Existuje také hodně zbytečného prostoru, kde jsou data shody inherentně duplikována přes úhlopříčku a většinu skutečné plochy grafu zabírá buď prázdné místo, nebo šum, a nakonec jsou bodové grafy omezeny na dvě sekvence. Žádné z těchto omezení se nevztahuje na zarovnávací diagramy Miropeats, ale mají své vlastní specifické nedostatky.
Bodové grafy lze také použít k posouzení opakovatelnosti v jedné sekvenci. Sekvence může být vynesena proti sobě a oblasti, které sdílejí významné podobnosti, se objeví jako čáry mimo hlavní úhlopříčku. K tomuto efektu může dojít, když se protein skládá z více podobných strukturálních domén .
Dynamické programování
Techniku dynamického programování lze použít k vytvoření globálních zarovnání pomocí algoritmu Needleman-Wunsch a lokálních zarovnání pomocí algoritmu Smith-Waterman . V typickém použití používají zarovnání proteinů substituční matici k přiřazení skóre k shodám aminokyselin nebo neshodám a penalizaci za mezeru za shodu aminokyseliny v jedné sekvenci s mezerou v druhé. Zarovnání DNA a RNA může používat bodovací matici, ale v praxi často jednoduše přiřadí kladné skóre shody, negativní skóre nesouladu a negativní mezeru. (Ve standardním dynamickém programování je skóre každé polohy aminokyseliny nezávislé na identitě jejích sousedů, a proto se efekty stohování bází neberou v úvahu. Je však možné tyto efekty zohlednit úpravou algoritmu.) A Společným rozšířením standardních nákladů na lineární mezeru je použití dvou různých sankcí za mezeru za otevření mezery a za prodloužení mezery. První je obvykle mnohem větší než druhý, např. -10 pro otevřenou mezeru a -2 pro prodloužení mezery. Počet mezer v zarovnání je tedy obvykle snížen a zbytky a mezery jsou drženy pohromadě, což obvykle dává větší biologický smysl. Algoritmus Gotoh implementuje náklady na afinitní mezeru pomocí tří matic.
Dynamické programování může být užitečné při srovnávání nukleotidů s proteinovými sekvencemi, což je úkol komplikovaný potřebou vzít v úvahu mutace posunu rámce (obvykle inzerce nebo delece). Metoda framesearch produkuje sérii globálních nebo lokálních párových zarovnání mezi vyhledávací nukleotidovou sekvencí a vyhledávací sadou proteinových sekvencí, nebo naopak. Jeho schopnost vyhodnotit posuny rámců kompenzované libovolným počtem nukleotidů činí metodu užitečnou pro sekvence obsahující velké množství indelů, což může být velmi obtížné sladit s efektivnějšími heuristickými metodami. V praxi tato metoda vyžaduje velké množství výpočetního výkonu nebo systém, jehož architektura je specializována na dynamické programování. Tyto BLAST a Reliéf apartmány poskytují základní nástroje pro tvorbu přeložené zarovnání (i když některé z nich se blíží využít vedlejších účinků vyhledány sekvence schopností nástrojů). Obecnější metody jsou dostupné z open-source softwaru , jako je GeneWise .
Je zaručeno, že metoda dynamického programování najde optimální zarovnání dané konkrétní bodovací funkce; identifikace dobré skórovací funkce je však často spíše empirická než teoretická záležitost. Přestože je dynamické programování rozšiřitelné na více než dvě sekvence, u velkého počtu sekvencí nebo extrémně dlouhých sekvencí je neúměrně pomalé.
Slovní metody
Wordové metody, známé také jako k -tuple metody, jsou heuristické metody, u nichž není zaručeno nalezení optimálního řešení zarovnání, ale jsou výrazně efektivnější než dynamické programování. Tyto metody jsou zvláště užitečné při rozsáhlém vyhledávání v databázi, kde je zřejmé, že velká část kandidátských sekvencí nebude mít v podstatě žádnou významnou shodu se sekvencí dotazu. Metody Word jsou nejlépe známé pro jejich implementaci v databázových vyhledávacích nástrojích FASTA a rodině BLAST . Metody Word identifikují řadu krátkých, nepřekrývajících se podsekvencí ("slov") v sekvenci dotazu, které jsou pak přiřazeny k sekvencím kandidátské databáze. Relativní polohy slova ve dvou porovnávaných sekvencích se odečtou, aby se získal posun; to bude indikovat oblast zarovnání, pokud více odlišných slov vytváří stejné odsazení. Pouze pokud je tato oblast detekována, aplikují tyto metody citlivější kritéria zarovnání; je tedy odstraněno mnoho zbytečných srovnání se sekvencemi bez znatelné podobnosti.
V metodě FASTA uživatel definuje hodnotu k, kterou má použít jako délku slova, pomocí které má prohledávat databázi. Metoda je pomalejší, ale citlivější při nižších hodnotách k , které jsou také upřednostňovány pro vyhledávání zahrnující velmi krátkou sekvenci dotazů. Rodina vyhledávacích metod BLAST poskytuje řadu algoritmů optimalizovaných pro konkrétní typy dotazů, jako je vyhledávání vzdáleně souvisejících sekvenčních shod. BLAST byl vyvinut tak, aby poskytoval rychlejší alternativu k FASTA, aniž by byla obětována velká přesnost; jako FASTA, BLAST používá vyhledávání slov o délce k , ale hodnotí pouze nejvýznamnější shody slov, nikoli každou shodu slov, jako FASTA. Většina implementací BLAST používá pevnou výchozí délku slova, která je optimalizována pro typ dotazu a databáze a která se mění pouze za zvláštních okolností, například při hledání s opakujícími se nebo velmi krátkými sekvencemi dotazů. Implementace lze nalézt na řadě webových portálů, jako jsou EMBL FASTA a NCBI BLAST .
Zarovnání více sekvencí

Zarovnání více sekvencí je rozšířením párového zarovnání tak, aby zahrnovalo více než dvě sekvence najednou. Více metod zarovnání se snaží zarovnat všechny sekvence v dané sadě dotazů. K identifikaci konzervovaných sekvenčních oblastí napříč skupinou sekvencí, u nichž se předpokládá, že jsou evolučně příbuzné, se často používá více zarovnání . Tyto konzervované sekvenční motivy mohou být použity ve spojení s konstrukčním a mechanistické informace lokalizovat katalytické aktivní místa z enzymů . Zarovnání se také používá k pomoci při navazování evolučních vztahů výstavbou fylogenetických stromů . Vyrovnání více sekvencí je výpočetně obtížné vyrobit a většina formulací problému vede k problémům kombinační optimalizace NP-Complete . Nicméně užitečnost těchto zarovnání v bioinformatice vedla k vývoji řady metod vhodných pro zarovnání tří nebo více sekvencí.
Dynamické programování
Technika dynamického programování je teoreticky použitelná pro libovolný počet sekvencí; protože je však výpočetně nákladný jak v čase, tak v paměti , používá se jen zřídka pro více než tři nebo čtyři sekvence v té nejzákladnější formě. Tato metoda vyžaduje konstrukci n -rozměrného ekvivalentu matice sekvence vytvořené ze dvou sekvencí, kde n je počet sekvencí v dotazu. Standardní dynamické programování se nejprve použije u všech párů sekvencí dotazů a poté se vyplní „zarovnávací prostor“ zvážením možných shod nebo mezer v mezipolohách, případně se vytvoří zarovnání v podstatě mezi každým zarovnáním dvou sekvencí. Přestože je tato technika výpočetně nákladná, její záruka globálního optimálního řešení je užitečná v případech, kdy je potřeba přesně zarovnat jen několik sekvencí. V softwarovém balíčku MSA byla implementována jedna metoda pro snížení výpočetních požadavků dynamického programování, která se spoléhá na objektivní funkci „součet párů“ .
Progresivní metody
Progresivní, hierarchické nebo stromové metody generují vícenásobné zarovnání sekvencí tak, že nejprve zarovnají nejpodobnější sekvence a poté do zarovnání přidávají postupně méně související sekvence nebo skupiny, dokud není do řešení začleněna celá sada dotazů. Počáteční strom popisující příbuznost sekvence je založen na párových srovnáních, která mohou zahrnovat heuristické metody párového zarovnání podobné FASTA . Výsledky progresivního zarovnání jsou závislé na volbě „nejpříbuznějších“ sekvencí, a proto mohou být citlivé na nepřesnosti v počátečních párových zarovnáních. Většina progresivních metod zarovnání více sekvencí navíc váží sekvence v sadě dotazů podle jejich příbuznosti, což snižuje pravděpodobnost špatného výběru počátečních sekvencí, a tím zlepšuje přesnost zarovnání.
Mnoho variací progresivní implementace Clustalu se používá pro zarovnání více sekvencí, konstrukci fylogenetických stromů a jako vstup pro predikci proteinové struktury . Pomalejší, ale přesnější varianta progresivní metody je známá jako T-Coffee .
Iterační metody
Iterační metody se pokoušejí zlepšit silnou závislost na přesnosti počátečních párových zarovnání, což je slabá stránka progresivních metod. Iterační metody optimalizují objektivní funkci na základě zvolené metody bodování zarovnání přiřazením počátečního globálního zarovnání a následným přeladěním podmnožin sekvencí. Přeskupené podmnožiny se pak samy zarovnají, aby vytvořily zarovnání více sekvencí další iterace. Jsou přezkoumány různé způsoby výběru podskupin sekvence a objektivní funkce.
Hledání motivu
Nalezení motivu, také známé jako profilová analýza, konstruuje globální vícenásobné zarovnání sekvencí, které se pokouší zarovnat krátké konzervované sekvenční motivy mezi sekvence v sadě dotazů. To se obvykle provádí tak, že se nejprve vytvoří obecné globální zarovnání více sekvencí, načež se vysoce konzervované oblasti izolují a použijí ke konstrukci sady profilových matic. Profilová matice pro každou konzervovanou oblast je uspořádána jako skórovací matice, ale její četnost pro každou aminokyselinu nebo nukleotid v každé poloze je odvozena spíše z distribuce charakteru konzervované oblasti než z obecnější empirické distribuce. Profilové matice se pak použijí k hledání dalších sekvencí pro výskyt motivu, který charakterizují. V případech, kdy původní datový soubor obsahoval malý počet sekvencí nebo pouze vysoce příbuzné sekvence, jsou přidány pseudoúčty k normalizaci distribuce znaků zastoupené v motivu.
Techniky inspirované informatikou
Na problém zarovnání více sekvencí byla také použita řada obecných optimalizačních algoritmů běžně používaných v informatice. Skryté Markovovy modely byly použity k vytvoření pravděpodobnostních skóre pro rodinu možných vícenásobných zarovnání sekvencí pro danou sadu dotazů; ačkoli rané metody založené na HMM vytvářely podmanivý výkon, pozdější aplikace je považovaly za obzvláště účinné při detekci vzdáleně souvisejících sekvencí, protože jsou méně citlivé na hluk vytvářený konzervativními nebo semikonzervativními substitucemi. Genetické algoritmy a simulované žíhání byly také použity při optimalizaci skóre vícenásobného seřazení sekvencí, jak bylo posouzeno skórovací funkcí, jako je metoda součtu párů. Více úplných podrobností a softwarových balíků najdete v hlavním článku zarovnání více sekvencí .
Tyto Burrows-Wheeler transformace byla úspěšně použita k rychlému vyrovnání krátké čtení v populárních nástrojů, jako jsou motýlek a BWA. Viz FM-index .
Strukturální vyrovnání
Strukturální zarovnání, která jsou obvykle specifická pro sekvence proteinů a někdy RNA, využívají informace o sekundární a terciární struktuře molekuly proteinu nebo RNA k pomoci při zarovnání sekvencí. Tyto metody mohou být použity pro dvě nebo více sekvencí a typicky vytvářejí lokální zarovnání; protože však závisí na dostupnosti strukturálních informací, lze je použít pouze pro sekvence, jejichž odpovídající struktury jsou známy (obvykle pomocí rentgenové krystalografie nebo NMR spektroskopie ). Protože struktura proteinu i RNA je evolučně konzervovanější než sekvence, strukturální uspořádání může být spolehlivější mezi sekvencemi, které jsou velmi vzdáleně příbuzné a které se natolik rozcházejí, že srovnání sekvencí nemůže spolehlivě detekovat jejich podobnost.
Strukturální zarovnání se používají jako "zlatý standard" při hodnocení zarovnání pro predikci struktury proteinů na základě homologie, protože výslovně srovnávají oblasti proteinové sekvence, které jsou strukturálně podobné, a nespoléhají se výhradně na informace o sekvenci. Při predikci struktury však nelze použít jasně strukturovaná zarovnání, protože cíl, který má být modelován, je alespoň jedna sekvence v sadě dotazů, pro kterou struktura není známa. Ukázalo se, že s ohledem na strukturální uspořádání mezi cílovou a templátovou sekvencí lze produkovat vysoce přesné modely cílové proteinové sekvence; hlavním kamenem úrazu v predikci struktury založené na homologii je produkce strukturně přesných zarovnání poskytujících pouze informace o sekvenci.
DALI
Metoda DALI, neboli zarovnání distanční matice , je metoda založená na fragmentech pro konstrukci strukturálních zarovnání na základě kontaktních podobností mezi postupnými hexapeptidy v dotazovacích sekvencích. Může generovat párová nebo vícenásobná zarovnání a identifikovat strukturální sousedy dotazové sekvence v Protein Data Bank (PDB). To bylo použito ke konstrukci FSSP strukturální srovnávací databáze (Fold klasifikace na základě uspořádání struktura-struktura proteinů, nebo rodiny strukturně podobných proteinů). Webový server DALI je přístupný na DALI a FSSP je umístěn v The Dali Database .
SSAP
SSAP (sekvenční program pro zarovnání struktury) je dynamická programovací metoda strukturálního zarovnání, která jako porovnávací body používá vektory atom-atom v prostoru struktury. Od svého původního popisu byla rozšířena o vícenásobná i párová zarovnání a byla použita při konstrukci hierarchické databázové klasifikace proteinových záhybů CATH (Class, Architecture, Topology, Homology). K databázi CATH lze přistupovat pomocí klasifikace proteinové struktury CATH .
Kombinatorické rozšíření
Metoda kombinatorického prodloužení strukturního zarovnání generuje párové strukturální zarovnání pomocí lokální geometrie k zarovnání krátkých fragmentů dvou analyzovaných proteinů a poté sestavuje tyto fragmenty do většího uspořádání. Na základě opatření, jako je střední kvadratická vzdálenost tuhého tělesa, vzdálenosti zbytků, lokální sekundární struktura a okolní environmentální rysy, jako je hydrofobicita sousedů zbytků , jsou generována lokální zarovnání zvaná „páry seřazených fragmentů“, která se používají k vytvoření matice podobnosti představující všechny možné strukturální zarovnání v rámci předdefinovaných mezních kritérií. Dráha z jednoho stavu struktury proteinu do druhého je pak sledována maticí prodloužením rostoucího zarovnání po jednom fragmentu. Optimální taková cesta definuje zarovnání kombinatorického prodloužení. Webový server implementující metodu a poskytující databázi párových zarovnání struktur v Protein Data Bank je umístěn na webových stránkách Combinatorial Extension .
Fylogenetická analýza
Fylogenetika a zarovnání sekvence jsou úzce příbuzná pole kvůli společné potřebě vyhodnocení příbuznosti sekvence. Oblast fylogenetiky široce využívá uspořádání sekvencí při konstrukci a interpretaci fylogenetických stromů , které se používají ke klasifikaci evolučních vztahů mezi homologními geny zastoupenými v genomech odlišných druhů. Míra, v níž se sekvence v sadě dotazů liší, kvalitativně souvisí s evoluční vzdáleností sekvencí od sebe navzájem. Zhruba řečeno, vysoká identita sekvence naznačuje, že dotyčné sekvence mají relativně mladého nejnovějšího společného předka , zatímco nízká identita naznačuje, že divergence je starodávnější. Tato aproximace, která odráží hypotézu „ molekulárních hodin “, že k extrapolaci uplynulého času lze použít zhruba konstantní rychlost evoluční změny od doby, kdy se dva geny poprvé rozcházely (tj. Doba koalescence ), předpokládá, že účinky mutace a selekce jsou konstantní napříč sekvenčními liniemi. Proto neodpovídá za možný rozdíl mezi organismy nebo druhy v rychlostech opravy DNA nebo možné funkční konzervaci specifických oblastí v sekvenci. (V případě nukleotidových sekvencí hypotéza molekulárních hodin ve své nejzákladnější formě také snižuje rozdíl v rychlosti přijetí mezi tichými mutacemi , které nemění význam daného kodonu, a jinými mutacemi, které vedou k začlenění jiné aminokyseliny do protein). Statisticky přesnější metody umožňují měnit rychlost evoluce na každé větvi fylogenetického stromu, a tak vytvářet lepší odhady doby koalescence pro geny.
Postupné techniky vícenásobného zarovnání nutně vytvářejí fylogenetický strom, protože do rostoucího zarovnání začleňují sekvence v pořadí příbuznosti. Jiné techniky, které sestavují vícenásobné zarovnání sekvencí a fylogenetické stromy, nejprve skórují a třídí stromy a vypočítávají zarovnání více sekvencí ze stromu s nejvyšším skóre. Běžně používané metody výstavby fylogenetických stromů jsou převážně heuristické, protože problém výběru optimálního stromu, stejně jako problém výběru optimálního vícenásobného zarovnání, je NP-tvrdý .
Posouzení významnosti
Seřazení sekvencí je užitečné v bioinformatice k identifikaci podobnosti sekvencí, produkci fylogenetických stromů a vývoji homologických modelů proteinových struktur. Biologický význam seřazení sekvencí však není vždy jasný. Uspořádání se často předpokládá, že odráží stupeň evoluční změny mezi sekvencemi pocházejícími od společného předka; je však formálně možné, že může dojít ke konvergentní evoluci za účelem vytvoření zjevné podobnosti mezi proteiny, které jsou evolučně nesouvisející, ale plní podobné funkce a mají podobné struktury.
Při vyhledávání v databázi, jako je BLAST, mohou statistické metody určit pravděpodobnost konkrétního zarovnání mezi sekvencemi nebo sekvenčními oblastmi, které vzniknou náhodou vzhledem k velikosti a složení prohledávané databáze. Tyto hodnoty se mohou výrazně lišit v závislosti na vyhledávacím prostoru. Zejména pravděpodobnost náhodného nalezení daného zarovnání se zvyšuje, pokud databáze obsahuje pouze sekvence ze stejného organismu jako sekvence dotazu. Opakující se sekvence v databázi nebo dotazu mohou také zkreslit výsledky vyhledávání i hodnocení statistické významnosti; BLAST automaticky filtruje takovéto opakující se sekvence v dotazu, aby se vyhnul zjevným zásahům, které jsou statistickými artefakty.
Metody odhadu statistické významnosti pro zarovnání mezerných sekvencí jsou dostupné v literatuře.
Posouzení důvěryhodnosti
Statistická významnost udává pravděpodobnost, že by zarovnání dané kvality mohlo nastat náhodou, ale neuvádí, o kolik je dané zarovnání lepší než alternativní zarovnání stejných sekvencí. Míry důvěryhodnosti zarovnání udávají, do jaké míry jsou nejlépe skórující zarovnání pro daný pár sekvencí v podstatě podobná. Metody odhadu důvěryhodnosti zarovnání pro zarovnání mezerných sekvencí jsou dostupné v literatuře.
Bodovací funkce
Volba skórovací funkce, která odráží biologická nebo statistická pozorování známých sekvencí, je důležitá pro dosažení dobrého zarovnání. Proteinové sekvence jsou často zarovnávány pomocí substitučních matic, které odrážejí pravděpodobnosti daných substitucí znak-znak. Série matic nazývaných PAM matice ( matice Point Accepted Mutation, původně definované Margaret Dayhoffovou a někdy označované jako „Dayhoffovy matice“) explicitně kódují evoluční aproximace týkající se rychlosti a pravděpodobnosti konkrétních aminokyselinových mutací. Další běžná řada bodovacích matic, známá jako BLOSUM (Blocks Substitution Matrix), kóduje empiricky odvozené pravděpodobnosti substituce. Varianty obou typů matic se používají k detekci sekvencí s různou úrovní divergence, což umožňuje uživatelům BLAST nebo FASTA omezit vyhledávání na těsněji související shody nebo rozšířit detekci divergentnějších sekvencí. Tresty za mezery představují zavedení mezery - v evolučním modelu, inzerční nebo deleční mutace - v sekvencích nukleotidů i proteinů, a proto by hodnoty trestů měly být úměrné očekávané rychlosti takových mutací. Kvalita vytvořených zarovnání tedy závisí na kvalitě bodovací funkce.
Může být velmi užitečné a poučné zkusit několikrát stejné zarovnání s různými možnostmi pro vyhodnocování hodnot trestu matice a/nebo mezery a porovnat výsledky. Regiony, kde je řešení slabé nebo nejedinečné, lze často identifikovat pozorováním, které oblasti zarovnání jsou odolné vůči změnám v parametrech zarovnání.
Jiná biologická použití
Sekvenovaná RNA, jako jsou exprimované sekvenční značky a mRNA o plné délce, může být zarovnána se sekvenovaným genomem, aby zjistila, kde jsou geny, a získala informace o alternativním sestřihu a editaci RNA . Sekvenční zarovnání je také součástí sestavy genomu , kde jsou sekvence zarovnány tak, aby se našlo překrytí, takže mohou být vytvořeny kontigy (dlouhé úseky sekvence). Dalším využitím je analýza SNP , kde jsou sekvence od různých jedinců zarovnány tak, aby nalezly jednotlivé základní páry, které se v populaci často liší.
Nelogické použití
Metody používané pro zarovnání biologických sekvencí našly uplatnění také v jiných oblastech, zejména ve zpracování přirozeného jazyka a ve společenských vědách, kde je Needleman-Wunschův algoritmus obvykle označován jako Optimální shoda . Techniky, které generují sadu prvků, ze kterých budou vybrána slova v algoritmech generování přirozeného jazyka, si vypůjčily více technik zarovnání sekvencí z bioinformatiky k výrobě lingvistických verzí počítačem generovaných matematických důkazů. V oblasti historické a srovnávací lingvistiky bylo sekvenční zarovnání použito k částečné automatizaci srovnávací metody , kterou lingvisté tradičně rekonstruují jazyky. Obchodní a marketingový výzkum také použil několik technik zarovnání sekvencí při analýze sérií nákupů v průběhu času.
Software
Úplnější seznam dostupného softwaru roztříděného podle algoritmů a typu zarovnání je k dispozici v softwaru pro zarovnání sekvence , ale mezi běžné softwarové nástroje používané pro obecné úlohy zarovnání sekvence patří ClustalW2 a T-coffee pro zarovnání a BLAST a FASTA3x pro vyhledávání v databázi. K dispozici jsou také komerční nástroje jako DNASTAR Lasergene , Geneious a PatternHunter . Nástroje označené jako provádějící zarovnání sekvence jsou uvedeny v registru bio.tools .
Algoritmy zarovnání a software lze vzájemně přímo porovnávat pomocí standardizované sady referenčních referenčních více sekvenčních zarovnání známých jako BAliBASE. Datový soubor se skládá ze strukturálních zarovnání, která lze považovat za standard, s nímž se porovnávají čistě sekvenční metody. Relativní výkon mnoha běžných metod zarovnání na často se vyskytující problémy se zarovnáním byl uveden do tabulky a vybrané výsledky byly publikovány online na BAliBASE. V proteinovém pracovním stole STRAP lze vypočítat komplexní seznam skóre BAliBASE pro mnoho (aktuálně 12) různých nástrojů pro zarovnání.
Viz také
- Sekvenční homologie
- Sekvenční těžba
- VÝBUCH
- Algoritmus pro vyhledávání řetězců
- Sekvenční analýza bez zarovnání
- UGENE
- Algoritmus Needleman – Wunsch
- Alogritmus Smith-Waterman
Reference
externí odkazy
-
 Média související se zarovnáním sekvencí na Wikimedia Commons
Média související se zarovnáním sekvencí na Wikimedia Commons