Environmentální DNA - Environmental DNA

| Část série na |
| DNA čárový kód |
|---|

|
| Podle taxonů |
| jiný |
Environmentální DNA nebo eDNA je DNA, která je shromažďována z různých environmentálních vzorků, jako je půda , mořská voda , sníh nebo dokonce vzduch, a nikoli přímo z jednotlivého organismu. Jak různé organismy interagují s prostředím, DNA je vyloučena a hromadí se v jejich okolí z různých zdrojů. Mezi příklady zdrojů eDNA patří, ale bez omezení na ně, výkaly , sliz , gamety , prolitá kůže, jatečně upravená těla a vlasy . Takové vzorky lze analyzovat pomocí vysoce výkonných sekvenačních metod DNA , známých jako metagenomika , metabarcoding a detekce jednoho druhu, za účelem rychlého monitorování a měření biologické rozmanitosti . Aby bylo možné lépe rozlišovat mezi organismy ve vzorku, používá se metabarcoding DNA, ve kterém je vzorek analyzován, a používá dříve studované knihovny DNA, jako je BLAST , k určení, jaké organismy jsou přítomny.
eDNA metabarcoding je nová metoda hodnocení biologické rozmanitosti, ve které jsou vzorky odebírány z prostředí pomocí vody, sedimentu nebo vzduchu, ze kterého je extrahována DNA, a poté amplifikovány pomocí obecných nebo univerzálních primerů v polymerázové řetězové reakci a sekvenovány pomocí sekvenování nové generace za účelem generování tisíců na miliony přečtení. Z těchto údajů lze určit přítomnost druhů a vyhodnotit celkovou biologickou rozmanitost. Jedná se o interdisciplinární metodu, která spojuje tradiční oborovou ekologii s hloubkovými molekulárními metodami a pokročilými výpočetními nástroji.
Analýza eDNA má velký potenciál nejen pro monitorování běžných druhů, ale také pro genetickou detekci a identifikaci dalších existujících druhů, které by mohly ovlivnit úsilí o zachování. Tato metoda umožňuje biomonitorování bez nutnosti shromažďování živého organismu, což vytváří schopnost studovat invazivní, nepolapitelné nebo ohrožené organismy, aniž by na organismus působil antropogenní stres. Přístup k těmto genetickým informacím zásadním způsobem přispívá k porozumění velikosti populace, distribuci druhů a populační dynamice u druhů, které nejsou dobře zdokumentovány. Důležité je, že eDNA je často nákladově efektivnější ve srovnání s tradičními metodami vzorkování. Integrita vzorků eDNA závisí na jeho zachování v prostředí.
Půda, permafrost , sladká voda a mořská voda jsou dobře prozkoumaná makroprostředí, ze kterých byly extrahovány vzorky eDNA, z nichž každé obsahuje mnohem více podmíněných subprostředí . Vzhledem ke své univerzálnosti je eDNA používána v mnoha dílčích prostředích, jako je odběr vzorků sladké vody, odběr vzorků mořské vody, odběr vzorků zemské půdy (tundra permafrost), odběr vzorků vodní půdy (řeky, jezera, rybníky a oceánské sedimenty) nebo v jiných prostředích, kde lze běžnými postupy odběru vzorků stát se problematickým.
Přehled
Environmentální DNA nebo eDNA popisuje genetický materiál přítomný ve vzorcích prostředí, jako je sediment, voda a vzduch, včetně celých buněk, extracelulární DNA a potenciálně celých organismů. eDNA lze zachytit ze vzorků prostředí a konzervovat, extrahovat, zesilovat, sekvenovat a kategorizovat na základě její sekvence. Z těchto informací je možná detekce a klasifikace druhů. eDNA může pocházet z kůže, sliznic, slin, spermií, sekretů, vajíček, výkalů, moči, krve, kořenů, listů, ovoce, pylu a tlejících těl větších organismů, zatímco mikroorganismy lze získat celé. Produkce eDNA závisí na biomase, věku a výživové aktivitě organismu, jakož i na fyziologii, historii života a využití prostoru.
Přestože jde o relativně novou metodu průzkumu, eDNA již prokázala obrovský potenciál v biologickém monitorování. Konvenční metody průzkumu bohatství a početnosti jsou omezeny taxonomickou identifikací, mohou způsobit narušení nebo zničení stanoviště a mohou se spoléhat na metody, ve kterých je obtížné detekovat malé nebo nepolapitelné druhy, což znemožňuje odhady pro celá společenství. eDNA může tyto metody doplnit zaměřením na různé druhy, vzorkováním větší diverzity a zvýšením taxonomického rozlišení . EDNA je navíc schopna detekovat vzácné druhy, ale nikoli určovat informace o kvalitě populace, jako jsou pohlaví a tělesné podmínky, takže je ideální pro doplnění tradičních studií. Bez ohledu na to má užitečné aplikace při detekci prvních výskytů invazivních druhů, pokračující přítomnosti původních druhů, které jsou považovány za vyhynulé nebo jinak ohrožené, a dalších nepolapitelných druhů vyskytujících se v nízké hustotě, kterou by bylo obtížné detekovat tradičními prostředky.
Degradace eDNA v prostředí omezuje rozsah studií eDNA, protože často zůstávají pouze malé segmenty genetického materiálu, zejména v teplých tropických oblastech. Navíc různě dlouhá doba do degradace na základě podmínek prostředí a potenciálu DNA cestovat po médiích, jako je voda, může ovlivnit odvozování časoprostorových trendů drobných druhů a společenstev. Navzdory těmto nevýhodám má eDNA stále potenciál určit relativní nebo hodnotnou hojnost, protože některé studie zjistily, že odpovídá biomase, ačkoli variabilita inherentní ve vzorcích životního prostředí ztěžuje kvantifikaci. Zatímco eDNA má mnoho aplikací v oblasti ochrany, monitorování a hodnocení ekosystémů, stejně jako další, které teprve budou popsány, vysoce variabilní koncentrace eDNA a potenciální heterogenita ve vodním útvaru činí nezbytným, aby byl postup optimalizován, ideálně s pilotní studií pro každou novou aplikaci zajistit, aby návrh vzorkování byl vhodný pro detekci cíle.
Komunitní DNA
Zatímco definice eDNA se zdá být přímočará, linie mezi různými formami DNA se stírají, zejména ve srovnání s komunitní DNA , která je popisována jako hromadné vzorky organismu. Nabízí se otázka ohledně celých mikroorganismů zachycených ve vzorcích eDNA: mění tyto organismy klasifikaci vzorku na společný vzorek DNA? Klasifikace genetického materiálu z výkalů je navíc problematická a často se označuje jako eDNA. Diferenciace mezi těmito dvěma je důležitá, protože komunitní DNA indikuje přítomnost organismu v určitém čase a místě, zatímco eDNA může pocházet z jiného místa, z výkalů dravců nebo z dřívější přítomnosti, nicméně toto rozlišení je často nemožné. EDNA však lze volně klasifikovat tak, že zahrnuje mnoho sektorů výzkumu biologické rozmanitosti DNA, včetně fekální analýzy a hromadných vzorků, pokud jsou použitelné pro výzkum biologické rozmanitosti a analýzu ekosystémů.
selfDNA
Koncept selfDNA vychází z objevů vědců z Univerzity v Neapoli Federica II , které byly v průběhu roku 2015 zaznamenány v časopise New Phytologist ( https://www.newphytologist.org/news/view/97 ), o inhibiční účinek extracelulární DNA u rostlin (), ale také u bakterií, hub, řas, rostlin, prvoků a hmyzu (). Navrhuje se, aby environmentálním zdrojem takové extracelulární DNA byl rostlinný odpad, ale také jiné zdroje v různých ekosystémech a organismech, přičemž velikost fragmentů DNA experimentálně prokázala inhibiční účinek na jejich konspecifické organismy se obvykle pohybuje v rozmezí 200 až 500 párů bází. Předpokládá se, že fenomén selfDNA řídí ekologické interakce a je mechanicky zprostředkován molekulárními vzory spojenými s poškozením (DAMP) (,) a má potenciál pro vývoj biocidních aplikací (). Viz také Cirkulující volná DNA .
eDNA metabarcoding
Do roku 2019 byly metody ve výzkumu eDNA rozšířeny, aby bylo možné hodnotit celé komunity z jednoho vzorku. Tento proces zahrnuje metabarcoding , který lze přesně definovat jako použití obecných nebo univerzálních primerů polymerázové řetězové reakce (PCR) na smíšených vzorcích DNA jakéhokoli původu následované vysoce výkonným sekvenováním příští generace (NGS) k určení druhového složení vzorek. Tato metoda je v mikrobiologii běžná již roky, ale nachází se pouze při hodnocení makroorganismů. Ekosystémové aplikace metabarcodingu eDNA mají potenciál nejen popisovat komunity a biologickou rozmanitost, ale také detekovat interakce a funkční ekologii ve velkých prostorových měřítcích, i když to může být omezeno falešnými údaji kvůli kontaminaci nebo jiným chybám. Metabarcoding eDNA celkově zvyšuje rychlost, přesnost a identifikaci oproti tradičnímu čárovému kódování a snižuje náklady, ale musí být standardizován a sjednocen a integrovat taxonomii a molekulární metody pro úplné ekologické studium.
Metabarcoding eDNA má aplikace pro monitorování rozmanitosti ve všech stanovištích a taxonomických skupinách, rekonstrukci starověkého ekosystému, interakce opylovačů rostlin , analýzu stravy, detekci invazivních druhů, reakce na znečištění a monitorování kvality ovzduší. Metabarcoding eDNA je jedinečnou metodou, která je stále ve vývoji a pravděpodobně zůstane v toku nějakou dobu, protože technologický pokrok a postupy se stanou standardizovanými. Vzhledem k tomu, že je metabarcoding optimalizován a jeho používání je stále rozšířenější, pravděpodobně se stane základním nástrojem ekologického monitorování a studie globální ochrany.
Extracelulární a reliktní DNA
Extracelulární DNA, někdy nazývaná reliktní DNA, je DNA z mrtvých mikrobů. Nahá extracelulární DNA (eDNA), jejíž většina se uvolňuje buněčnou smrtí, je v prostředí téměř všudypřítomná. Jeho koncentrace v půdě může být až 2 μg/l a jeho koncentrace v přírodním vodním prostředí může být až 88 μg/l. Pro eDNA byly navrženy různé možné funkce: může se podílet na horizontálním přenosu genů ; může poskytovat živiny; a může působit jako pufr pro nábor nebo titraci iontů nebo antibiotik. Extracelulární DNA působí jako funkční složka extracelulární matrice v biofilmech několika bakteriálních druhů. Může fungovat jako rozpoznávací faktor k regulaci připojení a rozptýlení konkrétních typů buněk v biofilmu; může přispívat k tvorbě biofilmu; a může přispět k fyzické síle biofilmu a odolnosti vůči biologickému stresu.
Pod názvem environmentální DNA zaznamenala eDNA zvýšené využití v přírodních vědách jako nástroj průzkumu ekologie , monitorování pohybů a přítomnosti druhů ve vodě, vzduchu nebo na souši a hodnocení biologické rozmanitosti oblasti.
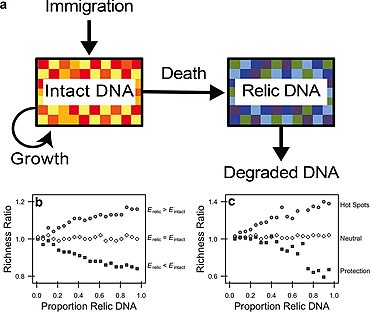
V diagramu vpravo je množství reliktní DNA v mikrobiálním prostředí určeno vstupy spojenými s úmrtností životaschopných jedinců s intaktní DNA a ztrátami spojenými s degradací reliktní DNA. Pokud je diverzita sekvencí obsažených ve fondu DNA relikvií dostatečně odlišná od rozmanitosti v intaktním souboru DNA, pak může DNA relikvií při odběru vzorků z celkové (intaktní + relikvie) DNA zkreslit odhady mikrobiální biodiverzity (jak ukazují různá barevná políčka). bazén. Standardizované údaje o iniciativách (STARDIT) byly navrženy jako jeden způsob standardizace jak údajů o metodách odběru a analýzy, tak o taxonomických a ontologických vztazích.
Environmentální RNA
Sbírka
Pozemské sedimenty
Důležitost analýzy eDNA pramení z uznání omezení, která představují studie založené na kultuře . Organismy se přizpůsobily, aby se jim dařilo ve specifických podmínkách jejich přirozeného prostředí. Ačkoli vědci pracují na napodobování těchto prostředí, mnoho mikrobiálních organismů nelze odstranit a kultivovat v laboratorním prostředí. Nejstarší verze této analýzy začala ribozomální RNA ( rRNA ) v mikrobech, aby lépe porozuměla mikrobům, kteří žijí v nepřátelském prostředí. Genetická výbava některých mikrobů je pak přístupná pouze prostřednictvím analýzy eDNA. Analytické techniky eDNA byly poprvé použity na suchozemské sedimenty poskytující DNA z vyhynulých i existujících savců, ptáků, hmyzu a rostlin. Vzorky extrahované z těchto pozemských sedimentů se běžně označují jako „sedimentární starodávná DNA“ ( seda DNA nebo špinavá DNA). Analýzu eDNA lze také použít ke studiu současných lesních společenstev včetně všeho od ptáků a savců po houby a červy. Vzorky lze získat z půdy, výkalů, „kousavé DNA“ z místa, kde byly kousány listy, z rostlin a listů, kde byla zvířata, a z krevních pokrmů ulovených komárů, kteří mohli sežrat krev jakéhokoli zvířete v dané oblasti. Některé metody se také mohou pokusit zachytit buňky pomocí pastí na vlasy a smirkového papíru v oblastech běžně překračovaných cílovými druhy.
Vodní sedimenty
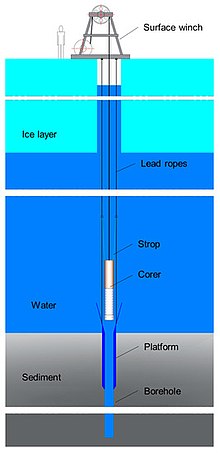
SedaDNA byla následně použita ke studiu starověké rozmanitosti zvířat a ověřena pomocí známých fosilních záznamů ve vodních sedimentech. Vodní sedimenty jsou zbaveny kyslíku a chrání tak DNA před degradací. Kromě starověkých studií lze tento přístup použít k pochopení současné rozmanitosti zvířat s relativně vysokou citlivostí. Zatímco u typických vzorků vody může dojít k poměrně rychlé degradaci DNA, vzorky vodního sedimentu mohou mít užitečnou DNA dva měsíce poté, co byl tento druh přítomen. Jedním z problémů vodních sedimentů je, že není známo, kde organismus uložil eDNA, protože by se mohl pohybovat ve vodním sloupci.
Vodní (vodní sloupec)
Studium eDNA ve vodním sloupci může naznačovat komunitní složení vodního útvaru. Před eDNA bylo hlavním způsobem studia rozmanitosti otevřené vody využívání rybolovu a chytání do pastí, což vyžaduje zdroje, jako je financování a kvalifikovaná pracovní síla, zatímco eDNA potřebuje pouze vzorky vody. Tato metoda je účinná, protože pH vody neovlivňuje DNA tak, jak se dříve myslelo, a citlivost lze relativně snadno zvýšit. Citlivost udává, jak pravděpodobný bude DNA marker ve vzorkované vodě, a lze ji zvýšit jednoduše odebráním více vzorků, většími vzorky a zvýšením PCR . eDNA se ve vodním sloupci poměrně rychle rozkládá, což je velmi výhodné v krátkodobých studiích ochrany, jako je identifikace přítomných druhů.
Vědci z oblasti experimentálních jezer v Ontariu v Kanadě a na McGill University zjistili, že distribuce eDNA odráží stratifikaci jezer . Jak se mění roční období a teplota vody, mění se také hustota vody tak, že v létě i v zimě vytváří v malých boreálních jezerech výrazné vrstvy . Tyto vrstvy se mísí na jaře a na podzim. Využití stanovišť ryb koreluje se stratifikací (např. Studenovodní ryby jako pstruh jezerní zůstanou ve studené vodě) a stejně tak distribuce eDNA, jak zjistili tito vědci.

V diagramu nahoře vlevo ukazuje růžová přerušovaná čára použití chemického indikátoru pro sledování kontaminace během odebírání jader. Bílá přerušovaná čára zobrazuje jádro sedimentu. Malé žluté kruhy označují teoretické intervaly vzorkování sedaDNA, odpovídající koláčovým grafům vpravo. Koláčové grafy představují hypotetická paleo-společenství detekovatelná analýzou brokovnice sedaDNA, kde většina (~ 75%) získaných sekvencí sedaDNA pochází z bakterií a kde se zvyšuje sedaDNA z fosilizujících/cystotvorných taxonů ve srovnání s nefosilizujícími/necystami -vytvářející taxony s hloubkou podmořského dna (za předpokladu, že sedaDNA fosilizujících/cystotvorných taxonů se zachová lépe než taxony nefosilizující/necystotvorné). Klesající velikost výsečových grafů s hloubkou podpodlaží naznačuje očekávaný pokles sedaDNA.
Sledování druhů
eDNA lze použít k monitorování druhů po celý rok a může být velmi užitečná při monitorování ochrany. Analýza eDNA byla úspěšná při identifikaci mnoha různých taxonů z vodních rostlin, vodních savců, ryb, mušlí, hub a dokonce i parazitů. eDNA byla použita ke studiu druhů při minimalizaci jakéhokoli stresu vyvolávajícího lidskou interakci, což vědcům umožňuje efektivněji sledovat přítomnost druhů ve větších prostorových měřítcích. Nejrozšířenějším využitím v současném výzkumu je použití eDNA ke studiu umístění ohrožených druhů, invazních druhů a klíčových druhů ve všech prostředích. eDNA je zvláště užitečná pro studium druhů s malými populacemi, protože eDNA je dostatečně citlivá na to, aby potvrdila přítomnost druhu s relativně malým úsilím sbírat data, což lze často provést pomocí vzorku půdy nebo vzorku vody. eDNA se spoléhá na účinnost genomového sekvenování a analýzy, jakož i na použité metody průzkumu, které jsou stále efektivnější a levnější. Některé studie ukázaly, že vzorky eDNA odebrané z proudu a pobřežního prostředí se rozpadly na nedetekovatelnou úroveň během přibližně 48 hodin.
Environmentální DNA lze použít jako nástroj k detekci organismů s nízkou četností v aktivní i pasivní formě. Aktivní průzkumy eDNA se zaměřují na jednotlivé druhy nebo skupiny taxonů pro detekci pomocí vysoce citlivých druhově specifických kvantitativních real-time PCR nebo digitálních kapkových markerů PCR . Metodika CRISPR-Cas byla také použita k detekci jednotlivých druhů z eDNA; s využitím enzymu Cas12a a umožňující větší specificitu při detekci sympatických taxonů. Pasivní průzkumy eDNA využívají masivně paralelní sekvenování DNA k amplifikaci všech molekul eDNA ve vzorku bez a priori cíle, což poskytuje plošný důkaz DNA o složení biotického společenství.
Pokles suchozemských členovců

Suchozemští členovci zažívají v Evropě i na celém světě masivní úbytek, přestože byl hodnocen pouze zlomek druhů a většina hmyzu je stále vědecky nepopsaná . Jako jeden příklad jsou ekosystémy travních porostů domovem různých taxonomických a funkčních skupin suchozemských členovců , jako jsou opylovači , phytofágní hmyz a dravci, kteří jako zdroj potravy používají nektar a pyl a pro potravu a vývoj stonkovou a listovou tkáň. Tato společenství chovají ohrožené druhy , protože mnoho stanovišť zmizelo nebo je výrazně ohroženo. Proto je vyvíjeno značné úsilí s cílem obnovit evropské ekosystémy travních porostů a zachovat biologickou rozmanitost . Například opylovači, jako jsou včely a motýli, představují důležitou ekologickou skupinu, která v Evropě prošla vážným poklesem, což naznačuje dramatický úbytek biologické rozmanitosti pastvin. Drtivá většina kvetoucích rostlin je opylována hmyzem a jinými zvířaty jak v mírných oblastech, tak v tropech. Většina druhů hmyzu jsou býložravci živící se různými částmi rostlin a většina z nich jsou specialisté, kteří jako hlavní zdroj potravy spoléhají na jeden nebo několik druhů rostlin. Vzhledem k mezerám ve znalostech o stávajících druzích hmyzu a skutečnosti, že většina druhů je stále nepopsaná, je zřejmé, že pro většinu rostlinných druhů na světě existují jen omezené znalosti o společenstvech členovců, které v sobě skrývají a interagují s nimi.
Společenstva suchozemských členovců byla tradičně shromažďována a studována pomocí metod, jako jsou past na malátnost a past na past , která je velmi účinná, ale poněkud těžkopádná a potenciálně invazivní. V některých případech tyto techniky nedosahují provádění efektivních a standardizovaných průzkumů, například kvůli fenotypové plasticitě , blízce příbuzným druhům a potížím s identifikací juvenilních stádií. Kromě toho, morfologická identifikace je přímo závislá na taxonomické odbornosti, který je v úpadku. Všechna taková omezení tradičního monitorování biologické rozmanitosti vyvolala poptávku po alternativních přístupech. Pokrok v technologiích sekvenování DNA mezitím nepřetržitě poskytuje nové způsoby získávání biologických dat. Proto bylo nedávno navrženo několik nových molekulárních přístupů pro získání rychlých a efektivních dat o společenstvech členovců a jejich interakcích prostřednictvím neinvazivních genetických technik. To zahrnuje extrakci DNA ze zdrojů, jako jsou velkoobjemové vzorky nebo hmyzí polévky, prázdné listové doly, pavučiny, tekutina z džbánů, vzorky životního prostředí, jako je půda a voda (environmentální DNA [eDNA]), identifikace hostitelské rostliny a dravé stravy z extraktů DNA hmyzu, a dravec scat z netopýrů. V poslední době se také DNA z pylu připojeného k hmyzu používá k získávání informací o interakcích rostlin a opylovačů . Mnoho z těchto nedávných studií spoléhá na metabarcoding DNA - sekvenování PCR amplikonů s vysokou propustností pomocí generických primerů.
Savci
Sněhové stopy
Výzkumníci divoké zvěře v zasněžených oblastech také používají vzorky sněhu ke shromažďování a získávání genetických informací o druzích zájmu. DNA ze vzorků sněhových stop byla použita k potvrzení přítomnosti takových nepolapitelných a vzácných druhů, jako jsou lední medvědi, polární liška, rys, rosomáci a rybáři.
DNA ze vzduchu
V roce 2021 vědci prokázali, že eDNA lze sbírat ze vzduchu a používat k identifikaci savců.
Řízení rybolovu
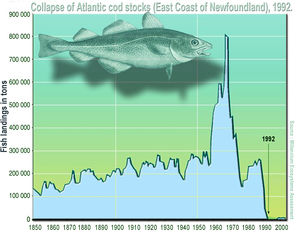
Úspěšné řízení komerčního rybolovu závisí na standardizovaných průzkumech pro odhad množství a rozdělení populací ryb . Treska obecná (Gadus morhua) je ikonickým příkladem, který ukazuje, jak špatně omezená data a neinformované rozhodování mohou mít za následek katastrofální pokles populace a následné ekonomické a sociální problémy. Tradiční hodnocení populace druhů žijících při dně se opíralo především o průzkumy vlečných sítí , které poskytly cenný proud informací osobám s rozhodovací pravomocí. Průzkumy vlečných sítí vlečných sítí pro lov při dně však mají určité významné nevýhody, včetně nákladů, selektivity/odlovu lovných zařízení, ničení stanovišť a omezeného pokrytí (např. Prostředí na dně s tvrdým substrátem, chráněné mořské oblasti).
Environmentální DNA (eDNA) se ukázala jako potenciálně účinná alternativa pro studium dynamiky ekosystémů. Neustálá ztráta a vylučování genetického materiálu z makroorganismů dodává molekulární stopu ve vzorcích životního prostředí, které lze analyzovat za účelem zjištění přítomnosti konkrétních cílových druhů nebo charakterizace biologické rozmanitosti. Kombinace sekvenování nové generace a vzorkování eDNA byla úspěšně použita ve vodních systémech k dokumentaci prostorových a časových vzorců v rozmanitosti rybí fauny. Pro další rozvoj využitelnosti eDNA pro řízení rybolovu je důležitým dalším krokem porozumění schopnosti množství eDNA odrážet biomasu ryb v oceánu.
V experimentálních systémech byl prokázán pozitivní vztah mezi množstvím eDNA a rybí biomasou a hojností. Očekává se však, že známé variace mezi produkcí eDNA a rychlostí degradace zkomplikují tyto vztahy v přírodních systémech. V oceánských systémech navíc velké objemy stanovišť a silné proudy pravděpodobně povedou k fyzickému rozptýlení fragmentů DNA mimo cílové organismy. Tyto matoucí faktory byly dříve považovány za omezení aplikace kvantitativního monitorování eDNA v oceánských podmínkách.
Navzdory těmto potenciálním omezením nalezly četné studie v mořském prostředí pozitivní vztahy mezi množstvím eDNA a úsilím o doplňkové průzkumy, včetně rádiového značení, vizuálních průzkumů, průzkumu ozvěny a průzkumu vlečných sítí. Studie, které kvantifikují cílové koncentrace eDNA komerčních druhů ryb se standardizovanými průzkumy vlečných sítí v mořském prostředí, jsou však mnohem vzácnější. V této souvislosti je nutné přímé srovnání koncentrací eDNA s metrikami pro hodnocení biomasy a populace, jako je úlovek na jednotku intenzity (CPUE), aby bylo možné porozumět použitelnosti monitorování eDNA, aby přispělo k úsilí v oblasti řízení rybolovu.
Hlubinné sedimenty
Extracelulární DNA v povrchových hlubinných sedimentech je zdaleka největším zásobníkem DNA světových oceánů. Hlavní zdroje extracelulární DNA v takových ekosystémech jsou reprezentovány uvolňováním DNA in situ z mrtvých bentických organismů a/nebo jinými procesy včetně buněčné lýzy způsobené virovou infekcí, buněčné exsudace a vylučování ze životaschopných buněk, rozkladu viru a alochtonních vstupů z vodní sloup. Předchozí studie poskytly důkaz, že důležitá část extracelulární DNA může uniknout degradačním procesům, přičemž zůstává zachována v sedimentech. Tato DNA potenciálně představuje genetické úložiště, které zaznamenává biologické procesy probíhající v průběhu času.
Nedávné výzkumy odhalily, že DNA konzervovaná v mořských sedimentech je charakterizována velkým počtem velmi rozmanitých genových sekvencí. Zejména extracelulární DNA byla použita k rekonstrukci minulé prokaryotické a eukaryotické rozmanitosti v bentických ekosystémech charakterizovaných nízkými teplotami a/nebo trvale anoxickými podmínkami.
Diagram vpravo ukazuje síť OTU ( operační taxonomická jednotka ) extracelulárních poolov DNA ze sedimentů různých kontinentálních okrajů. Velikost bodů v síti je úměrná množství sekvencí pro každou OTU. Body zakroužkované červeně představují extracelulární jádra OTU, tečka zakroužkovaná žlutě je částečně sdílená (mezi dvěma nebo více skupinami) OTU, tečky zakroužkované černě jsou OTU bez každého souboru. Jsou ukázány základní OTU přispívající alespoň pro 20 sekvencí. Čísla v závorkách představují počet připojení mezi OTU a vzorky: 1 pro exkluzivní OTU, 2–3 pro částečně sdílené OTU a 4 pro základní OTU.
Předchozí studie naznačovaly, že zachování DNA by mohlo být upřednostňováno také v bentických systémech charakterizovaných vysokými vstupy organické hmoty a rychlostí sedimentace, jako jsou kontinentální okraje. Tyto systémy, které představují ca. 15% globálního mořského dna jsou také hotspoty bentické prokaryotické rozmanitosti, a proto by mohly představovat optimální místa pro zkoumání prokaryotické diverzity zachované v extracelulární DNA.
Prostorová distribuce prokaryotické rozmanitosti byla intenzivně studována v bentických hlubinných ekosystémech prostřednictvím analýzy „environmentální DNA“ (tj. Genetického materiálu získaného přímo ze vzorků prostředí bez zjevných známek biologického zdrojového materiálu). Rozsah, v jakém mohou genové sekvence obsažené v extracelulární DNA změnit odhady diverzity dnešních prokaryotických sestav, není znám.
Sedimentární starověká DNA
|
−4500 -
-
-
-
−4000 -
-
-
-
−3500 -
-
-
-
−3000 -
-
-
-
−2500 -
-
-
-
−2000 -
-
-
-
−1500 -
-
-
-
−1000 -
-
-
-
−500 -
-
-
-
0 -
|
|
|
||||||||||||||||||||||||||||||||||||||||||
( před miliony let )
| ||||||||||||||||||||||||||||||||||||||||||||
Analýzy starověké DNA uchovávané v různých archivech změnily chápání evoluce druhů a ekosystémů. Zatímco dřívější studie se soustředily na DNA extrahovanou z taxonomicky omezených vzorků (jako jsou kosti nebo zmrazená tkáň), pokroky ve vysokovýkonném sekvenování a bioinformatice nyní umožňují analýzu staré DNA extrahované ze sedimentárních archivů, takzvané sedaDNA. Akumulace a uchování sedaDNA zakopané v pevninských a jezerních sedimentech byly předmětem aktivního výzkumu a interpretace. Studium depozice DNA na dně oceánu a její uchování v mořských sedimentech je však složitější, protože DNA musí cestovat několika kilometry vodním sloupcem . Na rozdíl od pozemského prostředí, s všudypřítomným transportem subfosilní biomasy z pevniny, je největší část mořské sedaDNA odvozena z planktonického společenství, kterému dominují mořští mikrobi a mořští protisté . Po smrti povrchového planktonu je jeho DNA vystavena transportu přes vodní sloupec, během kterého je známo, že velká část související organické hmoty je spotřebována a resp . Tento transport může trvat 3 až 12 dní v závislosti na velikosti a morfologii testu. Zůstává však nejasné, jak přesně planktonická eDNA, definovaná jako celková DNA přítomná v okolním prostředí, přežije tento transport, zda je degradace nebo transport spojen s tříděním nebo laterální advekcí a nakonec, zda eDNA přicházející na mořské dno je zachovány v mořských sedimentech bez dalšího narušení jejího složení.
Navzdory dlouhému vystavení degradaci za oxických podmínek během transportu ve vodním sloupci a podstatně nižší koncentraci organické hmoty na mořském dně existují důkazy, že planktonická eDNA je zachována v mořských sedimentech a obsahuje využitelný ekologický signál. Dřívější studie ukázaly uchování sedaDNA v mořských sedimentech uložených v anoxii s neobvykle vysokým množstvím konzervované organické hmoty, ale pozdější výzkumy ukazují, že sedaDNA lze také extrahovat z normálních mořských sedimentů, kde dominují klastické nebo biogenní minerální frakce. Nízká teplota hlubinné vody (0–4 ° C) navíc zajišťuje dobré uchování sedaDNA. Použití planktonických foraminifér jako „Rosettského kamene“, umožňujícího srovnávání podpisů sedaDNA souběžně se vyskytujícími fosilními testy těchto organismů, Morard et al. v roce 2017 ukázal, že otisk prstu planktonové eDNA přicházející na mořské dno zachovává ekologický podpis těchto organismů ve velkém geografickém měřítku. To naznačuje, že eDNA planktonové komunity je uložena na mořském dně níže, společně s agregáty, kostrami a dalším potápějícím se planktonickým materiálem. Pokud je to pravda, sedaDNA by měla být schopna zaznamenávat podpisy hydrografie povrchových oceánů, ovlivňující složení společenstev planktonu, se stejným prostorovým rozlišením jako kosterní pozůstatky planktonu. Pokud navíc planktonová eDNA přichází na mořské dno ve spojení s agregáty nebo skořápkami, je možné, že odolá transportu přes vodní sloupec fixací na minerální povrchy. Stejný mechanismus byl navržen k vysvětlení zachování sedaDNA v sedimentech, z čehož vyplývá, že tok planktonické eDNA zapouzdřené v kalcitovém testu přijíždějící na mořské dno je podmíněn uchováním při zakopání.
Planktonic foraminifera sedaDNA je ideálním zástupcem jak „horizontálně“ pro posouzení prostorového rozlišení rekonstrukce minulých hydrografických rysů povrchového oceánu, tak „vertikálně“, pro jednoznačné sledování pohřbení jeho signálu v koloně sedimentu. Tok eDNA planktonických foraminifer by měl být skutečně úměrný toku mrtvých foraminiferálních skořápek klesajících k mořskému dnu, což by umožňovalo nezávislé srovnávání signálu eDNA. eDNA je účinný nástroj ke studiu ekosystému, protože nevyžaduje přímé taxonomické znalosti, což umožňuje shromažďování informací o každém organismu přítomném ve vzorku, a to i na kryptické úrovni . Přiřazení sekvencí eDNA známým organismům se však provádí porovnáním s referenčními sekvencemi (nebo čárovými kódy ), které jsou k dispozici ve veřejných úložištích nebo v kurátorských databázích. Taxonomie planktonických foraminifer je dobře srozumitelná [1] </ref> a existují čárové kódy, které umožňují téměř úplné mapování eDNA amplikonů na taxonomii na základě morfologie foraminiferálních testů. Důležité je, že složení společenstev planktonických foraminifera je úzce spojeno s povrchovou hydrografií a tento signál je zachován fosilními testy uloženými na mořském dně. Protože foraminiferální eDNA nahromaděná v oceánském sedimentu lze získat zpět, mohla by být použita k analýze změn v planktonických a bentických společenstvech v průběhu času.
Participativní výzkum a občanská věda
Relativní jednoduchost vzorkování eDNA je vhodná pro projekty, které se snaží zapojit místní komunity do výzkumných projektů, včetně odběru a analýzy vzorků DNA. To může posílit místní komunity (včetně domorodých obyvatel), aby se aktivně zapojily do monitorování druhů v prostředí, a pomoci přijímat informovaná rozhodnutí v rámci modelu výzkumu participativní akce. Příklad takového projektu předvedla charitativní organizace Science for All s projektem „Wild DNA“.
Viz také
Reference
Další reference
- Schallenberg, Lena; Wood, Susie A .; Pochon, Xavier; Pearman, John K. (2020). „Co nám může DNA v prostředí říci o ekosystému?“ . Hranice pro mladé mysli . 7 . doi : 10,3389/frym.2019.00150 . S2CID 210714520 .